An overview of methods used in zebrafish research.
斑马鱼(Danio Rerio)是生活在印度、巴基斯坦或是其它亚洲国家河流中的一种淡水鱼。在过去的二十年中,斑马鱼已成为生物研究中一种常用的体内研究模式生物,尤其在胚胎发育、细胞迁移、器官发生,行为学以及睡眠研究等领域。近来斑马鱼也引发了生物医学研究领域其它分支的研究兴趣,这是因为斑马鱼具有成为多种疾病模型的潜力。本文总结了常用的斑马鱼研究方法。有关斑马鱼的饲养和操作方法在“斑马鱼手册” [8] 中已经有详细的描写,本文主要记述现有的斑马鱼研究的方法及其意义。
在过去的几十年中,斑马鱼已经成为最受欢迎的生物医学研究模型,研究方向涉及发育生物学、形态发生、神经科学、再生科学、衰老研究等等。易于饲养,后代繁育数量大,易于进行遗传筛选以及光透性的胚胎,是斑马鱼成为最佳动物模型的几个条件。尽管斑马鱼研究最重要的优点是可以得到高分辨率的体内图片,但跟果蝇(Drosophila melanogaster)和小鼠相比,由于缺乏合适的转基因工具来建立转基因动物以及进行基因敲除,斑马鱼研究相对落后。然而,在过去的几年中,这些空白已经被快速发展的科技手段所填补。诸如能用于高效基因敲除的CRISPR和锌指核酸酶或是MAZe斑马鱼品系以及脑彩虹转基因技术等新兴工具使得对复杂的发育过程进行评估以及理解脊椎动物疾病的发病机制成为了可能。 有关CRISPR应用于斑马鱼的研究已有优秀的综述和具体的实验步骤 [9-11] ,而且该技术正在迅速发展;因此本文不讨论CRISPR。
另外,人类和斑马鱼的基因组存在70%的同源性,提示有很大数量的基因和遗传途径存在保守性。现阶段可以利用的技术平台使得斑马鱼可以在完整生物体中用于中等通量的药筛(与无细胞系统或是单细胞系统相比)。本文重点着眼于相关方法,包括注射、免疫染色和原位标记,以及对基因表达下调、定向突变、转基因工具以及嵌合实验等进行了探讨。
最近的研究表明斑马鱼能感受的常用麻醉剂的MS-222,所以应该避免使用MS-222 [12, 13] 。
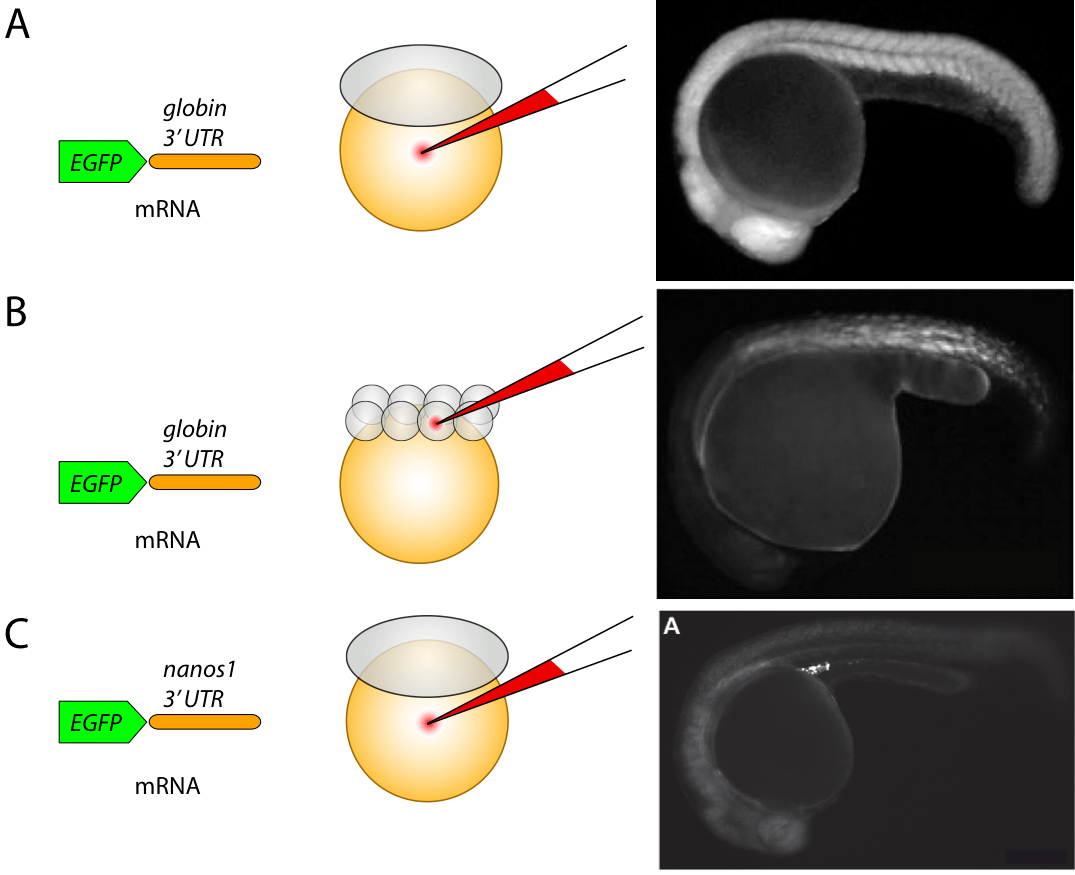
注射是斑马鱼研究中最常用的一项技术。通过注射把特定物质比如核酸、蛋白质或是特定药物引入胚胎中,这通常是在胚胎发育的早期阶段进行(图一)。注射通常是通过装载有所需注射物质的玻璃针进行。显微操纵器可以轻松地操纵注射针头在胚胎的不同位置进行注射。外接的特定装置可以通过压力将注射器装载的物质释放到胚胎中。这项技术可以实现物质的快速传输。有经验的研究人员在一小时内可以对几百个胚胎进行操作。注射说明书详见参考文献 [14] ,相关视频详见 [15] 。
显微注射最常用于胚胎特定蛋白的短期过表达。通过将编码目的蛋白的mRNA导入刚刚开始发育两小时的斑马鱼胚胎内,可以对斑马鱼胚胎的早期阶段(直到发育的第三天)进行特定蛋白的短期过表达。为了保持转入的mRNA转录本的稳定,3’端非翻译区(UTR)被加入到该转录本中(图一)。其中包含中的多聚A序列。为了驱动目的蛋白在发育胚胎中每个细胞中都表达,显微注射要在胚胎的单细胞时期进行(图一B)。而想要得到嵌合胚胎时,显微注射则应在稍后的8细胞胚胎中的某一个卵裂球中进行(图一A),从而使目的蛋白只在该卵裂球的子代细胞中表达。另外用于得到嵌合蛋白表达体的其它策略是通过把DNA质粒注射到单细胞阶段胚胎的单个卵裂球或者稍后阶段的胚胎中(直接注射到特定细胞),但这种方式效率稍低且跟mRNA注射相比蛋白合成所需的时间更长。
尽管通过mRNA注射来实现蛋白的短期表达可以快速简便地在斑马鱼早期发育过程中进行蛋白定位和功能研究,这种方法却不适合在发育晚期或是目的蛋白必须在特定组织和细胞中进行表达时使用。
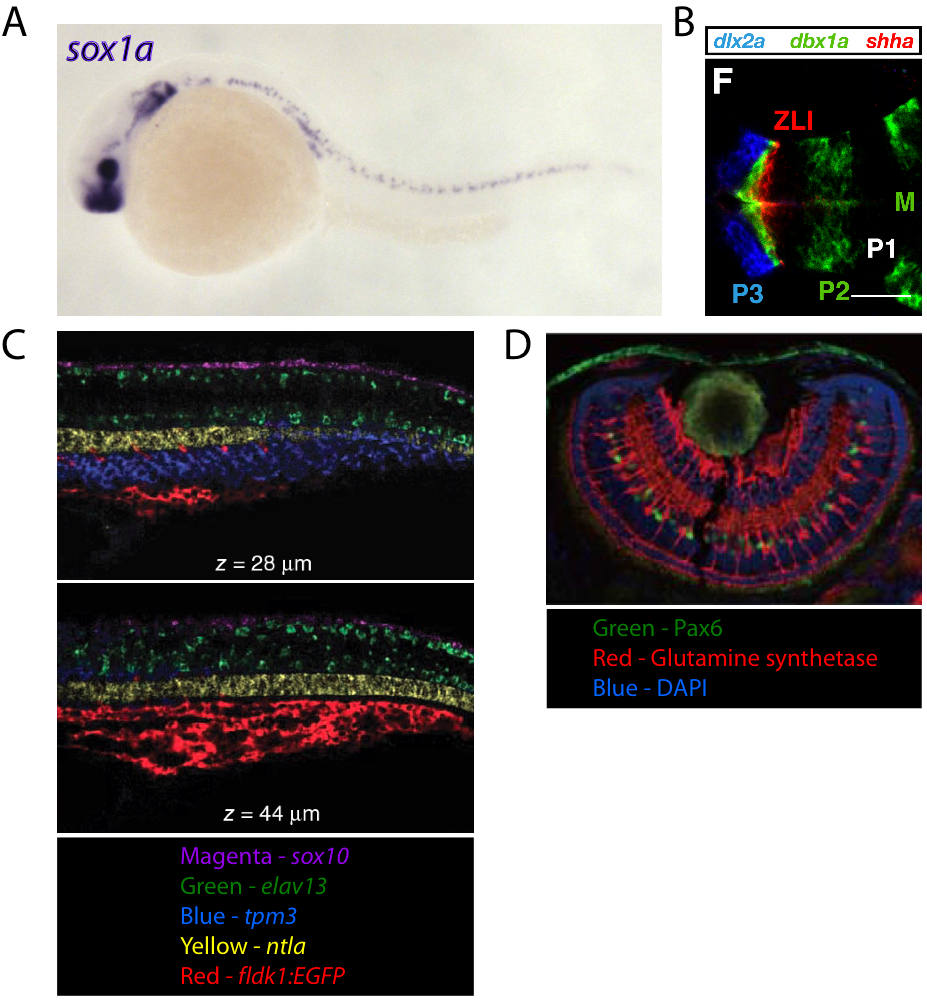
原位杂交技术是斑马鱼研究中最古老的方法之一。这项技术可以实现在完好的胚胎或是制作的切片标本 [16] 中研究基因表达情况。在原位杂交过程中,反义mRNA探针用于识别并结合内源转录本,随后通过着色分析或是荧光分析来进行探测。最经典的方式是通过着色标记探针,用光学显微镜(图2A)来检测信号。近来以荧光为基础的检测技术也已成功地优化并应用于斑马鱼研究中,使得能用常规方法同时检测三种探针或利用HCR扩增同时检测多达5个探针(图二B和C) [4, 17] 。荧光原位杂交以其高灵敏性和可以同时检测多个探针的优点成为了一种人们首选的信号检测的方法,而染色的方法只能同时检测两种探针。然而,传统的染色方法并不需要共聚焦显微镜。

免疫染色是一种有效的检测内源蛋白存在与否和存在位置的方法。在进行免疫染色过程中,最大的困难是抗原修复,另外目前适用于斑马鱼的高效商业化抗体也较少。最近有篇文章提出了一种改良的高效胚胎抗原修复的方法(图二D) [5] 。
现阶段已有许多转基因工具可以适用于斑马鱼和其它生物体,可以实现稳定的蛋白表达或是特定组织中目标蛋白的可调控表达。最常用的斑马鱼转基因系统是来源于青鳉的转座子体系 [18, 19] 。转基因结构中有两个来自Tol2元件的最小顺式调控序列分别位于最小启动子的5’和3’端,随后是荧光蛋白(图三)。To12载体最多可以容纳11Kb的DNA片段 [18] 。这种DNA结构与编码转座酶的mRNA编码序列一同被注射入单细胞阶段的胚胎细胞中(图三)。处理过的胚胎经培养后再从成鱼中筛选出在生殖细胞系中有插入片段并能传递到下一代的突变体。(如图三的F1所述)
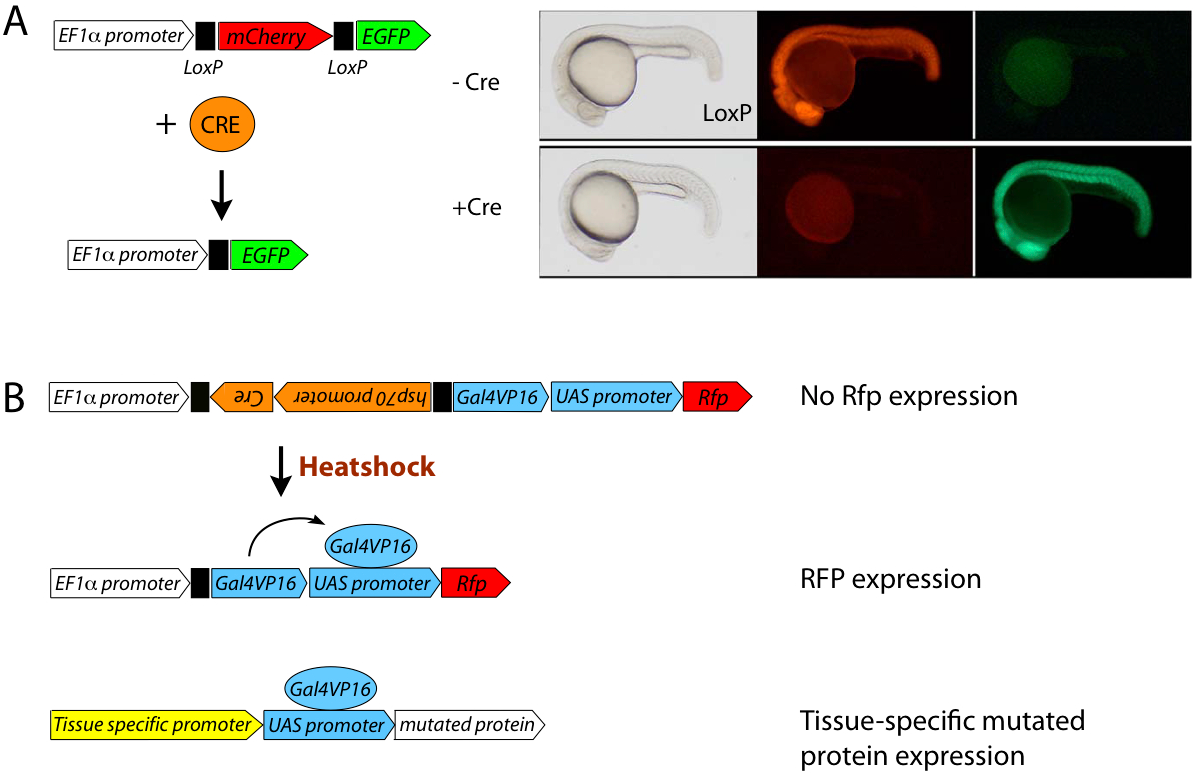
此外,近来人们发现可以通过cre介导的定向重组技术对斑马鱼的蛋白表达进行调控 [1] 。Cre介导的重组实现了具有时间可调控性的蛋白表达(图四A)。Cre重组表达可以通过热激启动子实现高精确性的时空特异性蛋白表达。这种策略已经被应用于MAZe转基因系统来实现嵌合胚胎中时空特异性蛋白达 [20] (图四B)。在热激启动子和GAL4/UAS系统的调控下应用MAZe系统与Cre-重组酶可以在斑马鱼体内进行表达研究。热激蛋白诱导生成Cre-重组酶,进而删除被lox P元件封闭的基因结构,随后表达的Gal4VP16蛋白可以与驱动目的蛋白表达的UAS启动子结合从而开启该蛋白的表达。这种方法非常适合在特定时间点特定细胞内诱导显性失活蛋白的表达。
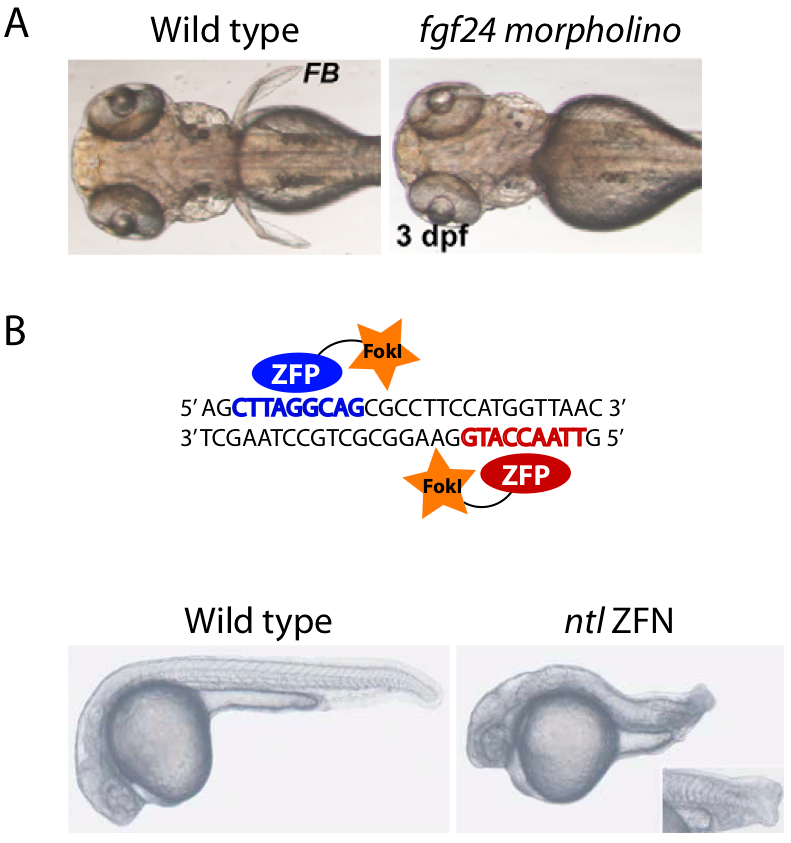
吗啉基寡核苷酸. 在斑马鱼胚胎的发育早期进行特定基因功能屏蔽的标准方法是利用一种修饰过的叫做吗啉基寡核苷酸的反义寡聚核苷酸链 [21, 22] 。吗啉基寡核苷酸是一个合成的分子,大约由25个核苷酸组成,可从基因工具一书中查到 [23] 。通常有两种类型的吗啉基寡核苷酸用以阻断蛋白表达:ATG 吗啉基寡核苷酸和拼接吗啉基寡核苷酸。其中ATG 吗啉基寡核苷酸通过在蛋白翻译的起始阶段作用于核糖体来实现胚胎发育中某种蛋白的缺失 [24] 。图五A显示的是一个经作用于fgf24基因的吗啉基寡核苷酸处理后的斑马鱼胚胎在发育到第3天时显示的胸鳍缺陷 [6] 。拼接吗啉基寡核苷酸可以对RNA拼接过程进行干扰,导致蛋白截短。因此,拼接吗啉基寡核苷酸可以用于研究特定蛋白的特定结构域功能。
近来人们又研发出了定向干扰内源microRNAs的靶保护者吗啉基寡核苷酸。这种吗啉基寡核苷酸的序列与miRNA结合靶mRNA的序列互补,从而可以阻止miRNA与其靶mRNA结合,进而稳定特定的mRNA转录本 [25] 。与此类似,吗啉基寡核苷酸也可以用于直接对miRNA的功能进行干扰 [26] 。在 [24, 27] 中作者概括论述了如何进行吗啉基寡核苷酸操作以及相关实验结果的确认原则。
尽管吗啉基注射是进行基因、miRNA干扰的一种高效率、高精确的策略,在我们进行吗啉基注射操作时还是要考虑到它的几点缺陷。最近研究表明,吗啉的效果并不特定;高达80%的观察到的吗啉诱导的表型是由于假阳性 [28] 。
- 通常吗啉基寡核苷酸都是被注射入卵黄中,会对发育中胚胎的所有细胞都产生同样的影响,当我们要干扰的蛋白在发育过程中广泛需要时就会危害到正常的胚胎发育。当然可以通过只把吗啉基注射到单个卵裂细胞(图一B)中来更精确地对特定细胞进行干扰。
- 注射入细胞中的吗啉基寡核苷酸只能对细胞持续作用数天(最高达 5天 [24] ),这就不利于干扰在发育晚期起作用的基因。
- 另外当细胞内存在其它同源蛋白时,吗啉基寡核苷酸往往不会起作用。
- 尽管吗啉基寡核苷酸可以对蛋白功能进行干扰,相应的mRNA还是存在于胚胎中。而当这种mRNA除了在蛋白翻译过程中作为模板外还有其它的作用,特别是当mRNA水平非常重要时,就可能会产生非特定效应。
锌指核酸酶。目前仍没有可靠的方法对斑马鱼进行定向突变,2008年发表的两篇文章描述了利用锌指酶(ZFN)在斑马鱼基因组中定点生成双链断裂。作者分别在两篇文章中介绍了利用ZFN来生成断裂点,经非同源末端连接修复后会发生小片段插入和删除。ZFN序列中包含有锌指蛋白(ZFP)序列和FokI内切酶位点 [29] (图五B和 [30, 31] )。在设计ZFN结构时,最重要的一点是优化ZFP对目的位点的识别性。在ZFIN网站 [32] 上面有详细的操作步骤和相关的背景知识,如何使用OPEN平台来设计锌指芯片在 [33] 有详细介绍。另外我们可以在 [34] 中查找ZFN作用位点的相关信息。
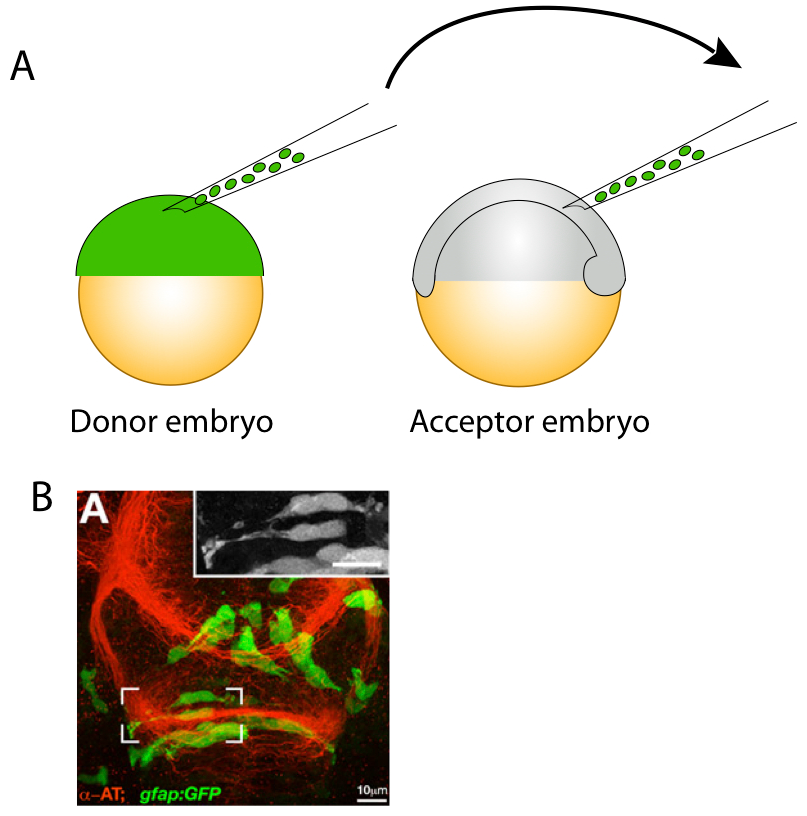
移植是发育生物学中应用得最古老的方法之一。在移植过程中,一团细胞从供体胚胎中移出,植入受体胚胎中(图六A)。移植技术对于解释许多发育生物学中的重要问题很重要,比如某基因的功能是否由其所在细胞决定。在研究基因功能时,突变细胞被移植到野生型胚胎中,相反的,野生型细胞则被移植到突变型胚胎中,进而对两种细胞及其宿主胚胎的表型进行比较。移植可以用于吗啉基寡核苷酸注射致死的情况。在这种情况下,可以通过从注射过吗啉基寡核苷酸的胚胎中移植细胞团到发育早期的野生型胚胎中,从而实现在野生型胚胎环境下研究敲除该蛋白后对该细胞所产生的影响。移植也用于对扩散因子效应的研究中,比如生长因子或是趋化因子。 在 [7, 35] (图六B)中展示的是一个利用移植的方法研究神经元-神经胶质间相互作用的例子。
- Ellingsen S, Laplante M, König M, Kikuta H, Furmanek T, Hoivik E, et al. Large-scale enhancer detection in the zebrafish genome. Development. 2005;132:3799-811 pubmed
- ZFIN ID: ZDB-IMAGE-070503-669. 来自: zfin.org/cgi-bin/webdriver?MIval=aa-imageview.apg&image_table=image&OID=ZDB-IMAGE-070503-669
- Manfroid I, Delporte F, Baudhuin A, Motte P, Neumann C, Voz M, et al. Reciprocal endoderm-mesoderm interactions mediated by fgf24 and fgf10 govern pancreas development. Development. 2007;134:4011-21 pubmed
- The Zebrafish Book. 来自: zfin.org/zf_info/zfbook/cont.html
- Microinjection of Zebrafish Embryos to Analyze Gene Function. 来自: www.jove.com/details.php?id=1115
- Jowett T, Lettice L. Whole-mount in situ hybridizations on zebrafish embryos using a mixture of digoxigenin- and fluorescein-labelled probes. Trends Genet. 1994;10:73-4 pubmed
- Kawakami K. Tol2: a versatile gene transfer vector in vertebrates. Genome Biol. 2007;8:S7 pubmed
- Summerton J. Morpholino antisense oligomers: the case for an RNase H-independent structural type. Biochim Biophys Acta. 1999;1489:141-58 pubmed
- Nasevicius A, Ekker S. Effective targeted gene 'knockdown' in zebrafish. Nat Genet. 2000;26:216-20 pubmed
- Gene Tools LLC. 来自: www.gene-tools.com/
- Choi W, Giraldez A, Schier A. Target protectors reveal dampening and balancing of Nodal agonist and antagonist by miR-430. Science. 2007;318:271-4 pubmed
- Flynt A, Li N, Thatcher E, Solnica-Krezel L, Patton J. Zebrafish miR-214 modulates Hedgehog signaling to specify muscle cell fate. Nat Genet. 2007;39:259-63 pubmed
- Porteus M, Carroll D. Gene targeting using zinc finger nucleases. Nat Biotechnol. 2005;23:967-73 pubmed
- Zinc Finger Consortium. 来自: zincfingers.org/default2.htm
- Tissue Targeted Embryonic Chimeras: Zebrafish Gastrula Cell Transplantation. 来自: www.jove.com/details.php?id=1422
- 来邦网
- 英文来邦
