An overview of antibody applications in biomedical research. Application keys used in Labome searches and webpages are explained.
| 应用 | 详情 | 来邦缩写 |
|---|---|---|
| 免疫印迹法 | 凝胶类型: 还原的, 天然的 裂解产物类型: 细胞裂解产物, 组织裂解产物, 免疫原过表达裂解产物 | WB |
| 单细胞免疫印迹法(single cell Western) | scWB | |
| 免疫组织化学 | 固定: 丙酮, 乙醇, 福尔马林/甲醛/多聚甲醛, 甲醇, 微波, 加热 包埋: 丙烯酸/塑料, 见如下 | IH |
| 包埋: 石蜡 | IHC-P | |
| 包埋: 冰冻 | IHC-F | |
| 自由漂浮切片法 | IHC-Free | |
| 免疫细胞化学 | IC | |
| 邻位连接技术 | PLA | |
| in-cell Western | ICW | |
| 流式细胞术 | FC | |
| 细胞分选 | FACS | |
| 免疫沉淀 | IP | |
| 染色质免疫沉淀 | ChIP | |
| 染色质免疫沉淀序列测定 | ChIP-Seq | |
| RNA免疫沉淀 | RIP | |
| 交联免疫沉淀 | CLIP | |
| 免疫测定 | ||
| ELISA | ELISA | |
| 放射性免疫测定 | RIA | |
| 酶联免疫测定 | EIA | |
| 免疫-PCR | I-PCR | |
| 功能测定 | ||
| 激活 | A | |
| 封闭/中和 | Neut | |
| 电泳迁移实验 | EMSA | |
| 质谱 | MS | |
| 抗肽抗体稳定同位素标准和捕获 | SISCAPA | |
| 免疫-MRM | I-MRM | |
| 免疫-MALDI | iMALDI | |
| 封闭/中和 | Neut | |
| 质谱免疫分析 | MSIA |
在生物医学研究领域,抗体是一类使用极其频繁的试剂。因其具有针对某种(些)特定表位的高敏感性和高特异性而成为研究应用中的理想试剂选择。此外,随着生物技术的不断发展进步,更加促进了抗体的大规模的生产和应用。在19世纪90年代,von Behring和Kitasato首先发现并描述了抗体。当时发现,非活性毒素能够在动物体内诱导出保护性免疫反应,从而对抗活性毒素,而且把这些产生了免疫保护的动物血清转移注射给其他动物,接受转移的动物也能获得同样的免疫保护。因此,抗体最初被称为抗毒素。但是,抗体后来才被发现其可以识别非常多种类的抗原。
根据最简单的定义,抗体即是B淋巴细胞抗原受体的可溶性形式,专门由成熟的B淋巴细胞产生。抗体免疫球蛋白分子由2条重链和2条轻链构成,根据其结构不同,可以把小鼠和人体内的抗体分为五类亚型。抗体分子的各个链之间以二硫键相连,从而使整个分子具有一定的灵活性。分子中没有轻链的部分称为可变区或Fc段(可结晶部分);此区域是由一组固定的基因所决定,同一个种属中的所有同亚型抗体此区域是一致的。分子中既有重链又有轻链的部分称为恒定区或Fab段(抗原结合区); 此区域的极端包含识别并结合靶抗原表位的高可变位点。Fab段也是由一组固定的基因决定,但需要进一步的体细胞突变产生独特的和高特异的可变位点(图1)。
目前是在动物体内或动物细胞内生产抗体。可从生产厂商购买或通过实验自行制备获得单克隆抗体(同种类亚型和抗原特异性)和多克隆(不同种类亚型和抗原特异性)抗体。多克隆抗体从被免疫靶抗原的动物体内的血清分离获得。而多克隆抗体是针对某一特定抗原中不同表位的不同抗体的混合物,因此也有可能包含针对无关蛋白的抗体,这可能会影响测定结果。 此外,多克隆抗体的产生依赖于其来源动物,即使针对的是同一种抗原,也没有2个生产批次的多克隆抗体是完全相同的。相反,单克隆抗体来源于杂交瘤细胞,这是一类永生的克隆细胞系,由小鼠或其他物种的B淋巴细胞与骨髓瘤细胞融合而成。此类细胞可以源源不断地产生完全相同的特异性抗体,这即是现在生产抗体的标准技术。
| 方法 | 数量 |
|---|---|
| 免疫印迹法 | 6351 |
| 免疫组织化学法 | 2898 |
| 免疫细胞化学 | 1969 |
| 免疫沉淀 | 1746 |
| 流式细胞术 | 772 |
| ELISA | 469 |
| 阻断或激活 | 355 |
| 染色质免疫沉淀 | 414 |
| 电泳迁移实验 | 277 |
免疫沉淀(IP),亦称为“pull-down”实验,即抗体在细胞裂解液, 生物体液, 或其它溶液中标记和沉淀(“pull-down”)分离出相应的目标抗原。通过在抗体的Fc段结合不同类别微珠(例如磁性珠,琼脂糖,琼脂糖凝胶),再用离心或其他机械方法把抗体抗原复合物从总蛋白中分离出来(图2)。
IP检测在许多细胞和分子生物学研究很受欢迎。根据其最基本的原理,IP常用来纯化目标抗原以进行后续研究。IP也常用于研究稳态细胞或经过特定处理的细胞中的各种蛋白质之间的相互作用。在通过IP研究蛋白质相互作用时,其中一个已知蛋白的抗体将用于免疫沉淀步骤,随后再可通过如免疫印迹法(见下文)来确定与这个已知蛋白结合并共同沉淀分离出来的另一种蛋白分子。
酶联免疫吸附试验(ELISA)用来对一个特定的可溶性蛋白分子进行定性和定量分析,如血清, 或液体的样品,如生物体液和细胞培养物上清液中的特异性抗体或蛋白质的有无及浓度。此方法是利用了聚苯乙烯检测板吸附蛋白质或抗体的能力及抗体特异性结合靶抗原的能力。一般来说,此方法可通过检测实验样品在一定波长下的吸光度,再结合已知的抗原或抗体浓度标准曲线,计算出检测样品中此抗原或抗体的浓度。有3种常用的ELISA检测方法。
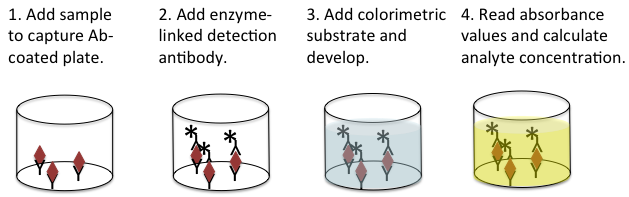
直接酶联试验使用不同量化的单克隆抗体来确定溶液中的某种特定抗原的浓度。 例如,直接ELISA可通过测量细胞培养上清中的细胞因子含量从而确定此群细胞(经刺激或抑制处理)分泌细胞因子的水平。直接ELISA法常以“三明治”法进行,即两种单克隆抗体通过不同的抗原表位结合在同一个靶抗原上,把靶抗原夹在中间。首先把“捕获”抗体包被在测定板上。然后在洗涤和封闭后,将待测溶液加入到测定孔中,让“捕获”抗体去捕获溶液中存在的靶抗原。洗去样品中其余的蛋白分子,在测定孔中加入偶联生物素“检测”抗体。再加入链霉抗生物素蛋白复合酶,以结合偶联生物素的检测抗体。最后,在测定孔中加入酶的显色底物。可以根据测定测定孔中的特定颜色变化进行相对定量: 颜色变化的越多,样品中的抗原浓度越高。可以通过已知靶抗原浓度的样品测定孔制作标准曲线,从而对未知浓度的样品测定孔进行绝对定量。这个检测过程不需要“捕获抗体”也可以进行;换句话说,抗原可直接包被到检测孔进行后续检测。但是,通过双抗体夹心法检测靶抗原其特异性更高(图3)。
间接酶联试验是用来检测溶液中的抗原特异性抗体的含量。在此项检测中,某种特定的抗原会直接包被到检测板的样品孔中。可能含有此种抗原特异性抗体的待检样品(如血清,杂交瘤细胞培养基)被加入到检测孔,特异性抗体抗原会相互结合。接下来,在检测孔加入可以结合抗体Fc段的酶联第二抗体,让它与已经结合在特异性抗原上的抗体相结合。最后,再加入比色底物,底物被第二抗体上偶联的酶酶解,通过吸光度的测定值反映所要检测抗体的含量。
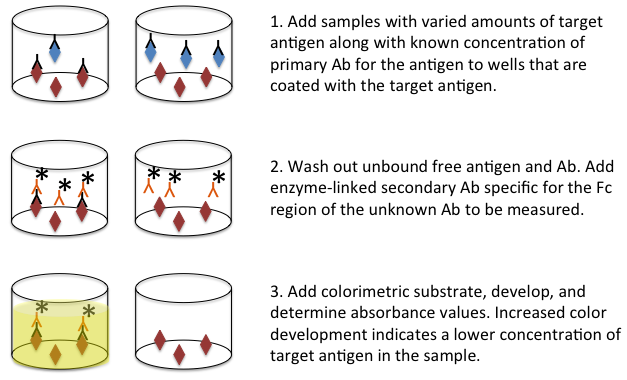
竞争性酶联试验通常用来检测小抗原分子。在此类检测中,把未知浓度的靶抗原样本加入到已包被了已知浓度的相同靶抗原的检测孔中。然后加入检测靶抗原的标记抗体。然后洗去可溶性的抗原抗体复合物,结合在检测孔中抗原上的抗体含量最后被检测到。竞争性ELISA法最后检测到的信号强度一般与其他ELISA方法相反。具体说来,样品中游离的抗原浓度越高,就会结合越多的抗体,因此结合到检测板上固定抗原的抗体就会减少,因此最后检测到的信号就会越弱(图4)。
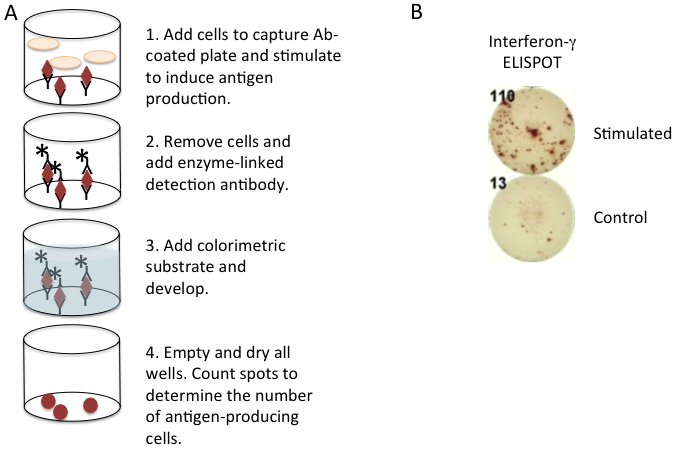
酶联斑点类似于夹心ELISA法检测细胞因子或其他可溶性因子,是通过结合在检测板上的单克隆抗体来捕获把抗原,以及可以用于进行比色测定的酶联第二单克隆抗体检测捕获的靶抗原。但是不同的是,在酶联免疫斑点检测中,产细胞因子的细胞可直接培养在预先包被了捕获抗体和封闭过的检测板中,此检测可以从一个斑点中捕获单个细胞所分泌的细胞因子。培养特定的一段后,弃去检测板中的细胞。其余后续的检测步骤同普通ELISA法非常类似。只是最后一步不是通过检测某波长下的吸光度来分析,而在显微镜直接进行视觉观察或使用专门的读板器。成功检测产细胞因子细胞的ELISPOT法在每个检测孔中会出现很多颜色深浅不一的斑点,每个斑点对应一个单细胞。ELISPOT法特别适用于检测细胞数量非常少的细胞群, 比如检测免疫小鼠的抗原特异性T细胞,此类数量非常少的细胞群用其他的方法一般很难检测到(图5)。
免疫吸附技术可与微阵列技术结合使用,从而实现高通量功能蛋白质组学测定。在此类检测中,预先在玻璃或聚苯乙烯载玻片上包被捕获抗体或样品(如细胞裂解产物)。前者(包被捕获抗体)类似的经典的夹心ELISA、ELISPOT,因为靶抗原结合在固定抗体和游离抗体之间,后者(直接包被样品)类似于直接ELISA法,因为靶抗原直接结合在载玻片上。这两种实验方法中的检测抗体都是针对于各种已知抗原的荧光标记抗体。该技术可快速筛选出表达或活化发生变化的蛋白质。
免疫PCR(I-PCR)结合了PCR核酸扩增的灵敏度,和基于抗体测定法的特异性,因此这项技术增加了检测灵敏度。通常情况下,可以在不同的应用中获得比ELISA方法的检测极限100-10000倍扩增。 Ⅰ-PCR最先在1992年由佐野等人所述 [1] 。尽管该技术有潜力,几年来在诊断和生物医学应用中I-PCR方法的用途被一些问题所限制,包括测试的持续时间,由此导致的错误率的增加。另外,主要的缺点之一是抗体难以与寡核苷酸结合。几种解决方案已经提出,如快速,高效的结合方法(如Innova Biosciences公司的Thunder-Link®抗体-寡核苷酸结合系统)。此外,多重和高通量技术的引入大大改善了I-PCR方法,这目前已用作一些细菌和病毒病原体的常规诊断测试,并用于检测肿瘤标志物和食品污染 [2] 。定量PCR与I-PCR相结合(QI-PCR),可以定量复杂生物样品中的低丰度生物标记物,这难以通过传统的免疫方法定量 [3] 。
蛋白质印迹是一种通过凝胶电泳分离蛋白质并通过电转移把蛋白质转移到吸附膜上。一经蛋白印迹后,即可通过特异性的标记抗体检测到相应蛋白质。
十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)可用以检测已被SDS化学变性的蛋白。然而,某些特定的抗体不会再一个变性的蛋白质分子上识别其抗原表位,因此需要进行不含SDS的聚丙烯酰胺凝胶电泳。
蛋白印迹可以通过湿法, 半干法, 或干法进行。在湿法,凝胶夹在印迹膜和各种过滤装置中间,然后把整个装置埋进一个装满三甘氨酸转移缓冲的电极槽中。在半干法,夹在中间的凝胶周围只有少量的缓冲,然后封闭在电极板之间。最后,在干式系统中不需要缓冲液,使用带有电极,缓冲液和膜的预组装即用的装置。 虽然干法转膜较快,但需要很好地拟合各部分的夹层配件,而湿法转膜很少会出现问题,可以尽量保持重复实验结果的一致性。此外,如果半干法转膜时间过长,较小分子量的蛋白质可能会从膜上转过。
转膜后,需对膜进行封闭,以减少非特异性蛋白结合,然后进行用一抗孵育膜,以研究感兴趣的目标蛋白。单克隆抗体通常具有更高的特异性;然而许多单克隆抗体产生于某个特定肽段而不是一个天然蛋白质,因此可能无法检测到PAGE所分离的天然蛋白。也可使用多克隆抗体,但易形成较高的背景值。因一抗通常无标记,所以需要一个可以结合在一抗Fc段的标记的二抗,以用于最后的检测。通常会用酶来标记二抗。酶标记后的印迹可通过化学发光酶底物及放射自显影技术成像。被抗体检测和标记到的蛋白质会出现在图像的暗区。
点印迹类似于免疫印迹法,在膜上检测目标蛋白;但是斑点印迹法中检测的蛋白质不用电泳进行分离。取而代之的是,含有目标蛋白的样品被直接“点”到膜上。这不是一种定量的方法,但它可以直接检测某种特定蛋白的有无; 例如,斑点印迹法可用于检测蔗糖梯度分离的细胞裂解产物中的某些蛋白质定位。
CHIP法有个局限性,即交联步骤可能会破坏蛋白分子结构,从而阻碍抗体与其结合,影响后续的免疫沉淀。此时,可以尝试无交联步骤的CHIP法;此过程称为N-ChIP的免疫沉淀法 [4, 6] 。尽管去交联提高抗原识别能力,但是一般只适用于目标蛋白强烈地结合在DNA上这种情况。
最近,芯片被改良以用于高通量分析。例如,ChIP-on-Chip结合了CHIP法和微阵列技术,适用于荧光标记序列的全基因组筛选。在这些检测中,用不同的荧光染料标记沉淀组DNA和对照组DNA(通常不经沉淀的总输入),然后用各组DNA与DNA微阵列芯片甚至整个基因组小寡核苷酸进行杂交。通过标准化分析技术,可以从芯片获得CHIP样品相对于DNA对照组的DNA结合位点的详细信息。
将染色质沉淀技术与现代高通量测序技术相结合,从而有利于鉴定前所未知的靶基因序列。由于高通量测序可以鉴定和提供大量的基因组信息,因此染色质免疫共沉淀测序适用于整个基因组的染色质免疫沉淀检测 [7] 。这个方法是一个非常强大的工具,可以鉴定全基因组DNA与蛋白质如转录因子的结合位点,从而揭示生物过程和基因调控。近日,一个以质谱为基础的称为内源性蛋白快速免疫质谱的方法(RIME)被提议与Chip-seq相结合。这种组合方法提供了目标蛋白的顺反组和相互作用组两种信息 [8] 。
交联免疫沉淀(CLIP)是首先由邮乐等人在2003年开发的方法。他们研究剪接因子NOVA和神经元特异性RNA结合蛋白(RBP)之间的相互作用 [9] 。类似于染色质免疫沉淀技术用来分析DNA-蛋白质的相互作用,交联免疫沉淀技术可用于分析RNA-蛋白质的相互作用 [10] 。染色质免疫共沉淀测序的方法的类似于染色质免疫沉淀;然而,因为目标核酸不同,因此也存在几个明显的差异。在染色质免疫共沉淀测序中,推荐使用的交联剂是紫外线照射。由于转录组RNA一般较短,所以不需要经过超声处理,细胞可以在标准缓冲液中裂解。但是必须在所有的裂解产物中添加RNase抑制剂以防止目标RNA的降解 [11] 。CLIP可以分析RNA-蛋白质相互作用,绘制全基因组规模的 [12] RNA结合位点。 尤其是HITS-CLIP已广泛用于绘制几个剪接因子,如PTB [13] ,FOX2 [14] ,以及Argonaute [15] 的蛋白质-RNA相互作用位点。然而,HITS-CLIP显现了与交联效率和RBP结合位点的精确测定相关的一些缺点。为了克服这些问题,在2010年哈夫纳等人开发了光活化 - 核糖核苷增强交联和免疫沉淀(PAR-CLIP) [16] ,在2011年柯尼希等提出了个别核苷酸方案CLIP(iCLIP),提供了RBP结合位点在单核苷酸水平的 解决方案 [17] 。
RNA免疫沉淀(RIP)是类似ChiP的方法,定性蛋白质和特定的RNA序列之间的相互作用 [18] 。主要步骤包括:i)在活细胞加入甲醛形成可逆的蛋白质-RNA交联; ⅱ)通过使用特异性抗体免疫沉淀目标蛋白质; ⅲ)交联的逆转; ⅳ)回收和用RT-PCR分析相关的RNA。 RIP可以与微阵列(RIP片)相关联,如在2006年 [19] 首述,或NGS技术(RIP-SEQ),从而可以通过几个过程如剪接,编辑,稳定,和RNA的翻译快速,准确的深入分析RNA和RBP,基因表达调控的关键要素。
电泳迁移率实验(EMSAs)是用来研究DNA结合蛋白对特定DNA位点的亲和力 [20] 。在经典的EMSAs中,含有结合位点的放射性标记的DNA片段与目标蛋白共同孵育。结合的亲和力是根据凝胶电泳中DNA向上迁移的速度来确定,由于结合了蛋白质的DNA,分子量发生了增加,从而导致在凝胶电泳中的迁移变缓慢。为了提高检测的特异性,可以在反应体系中加入目标蛋白的特异性抗体;如果抗体和目标蛋白结合,进一步减慢DNA-蛋白质复合物在凝胶电泳中的迁移速度,超级电泳迁移实验 [21] 。
免疫组织化学(IHC)和免疫细胞化学(ICC)是常用来在原位检测组织和细胞内蛋白表达和定位的两种方法。两种方法主要在样品制备上有所不同;IHC是在固定的整个组织切片上进行,而ICC是在从基质中分离出来的细胞中进行(例如,血液细胞可通过细胞离心涂片法,培养的细胞可在玻片上的长成单细胞层)(图8)。

免疫细胞化学的一个变化是在细胞免疫印迹法(in-cell Western: ICW) [22] 。在ICW实验中,长在多孔板细胞需经过和免疫细胞化学相同方式的固定处理, 然后和一抗反应。 一抗或二抗通常与近红外荧光分子轭联 (如DyLight® 680或800),以避免细胞和多孔板的自发荧光。每个孔中的信号再与另一具有不同的发射波长的近红外染料如DRAQ5 [23, 24] 进行标准化。ICW仅可以使用已被免疫细胞化学验证的抗体(注意:不是免疫印迹), 这样才能确保抗体和检测的特异性。
用于IHC或ICC技术的有几种常见的组织或细胞固定方法,固定方法的选择取决于检测分析的类型。用免疫组织化学检测的完整组织样本固定通常用甲醛,这是一种半可逆交联剂,来源于多聚甲醛,也可进一步稀释为福尔马林。虽有研究指出,在甲醛中过分固定会对样品产生负面影响,但其他也有报道称,并无负面影响,反而强调没有充分的固定时间将影响甲醛交联细胞蛋白质。用甲醛固定组织样本甚至是整个动物样本,是将其完全浸没至体积比为4%的工作浓度的甲醛溶液中去。
醇类,特别是甲醇和乙醇,经常用于免疫细胞化学技术中的细胞固定,因为它们不会损伤细胞中的DNA。醇类一般不推荐用于固体组织标本,因为不能像甲醛那样可以保留组织形态。此外,用醇类固定可导致组织发生收缩。丙酮不是常用的固定剂,一般只是用于快速冷冻组织的固定,因为它改善表位的检测,也因此用于甲醇固定后的第二步固定。但是丙酮固定也能导致组织收缩。最后需要指出,在所有的固定方法中蛋白抗原性的保存必不可少,组织标本可以保存在液态氮冷却过的异戊烷中,并存储在80ºC直到后续处理。
| 方法 | 使用范围 | 利与弊 |
|---|---|---|
| 甲醛类 | 完整组织 |
|
| 醇类 | 细胞(免疫细胞化学) |
|
| 丙酮 | 冰冻组织或甲醇固定后的再固定 |
|
| 快速冷冻 | 需要保存抗原表位 |
|
组织在切片和染色前,必须先包埋到某种基质中。石蜡是常用来包埋组织的。由于石蜡是疏水的,所以组织必须在一系列浓度梯度增高的乙醇中浸泡进行脱水。在灌注石蜡前的倒数第二步是把组织标本浸泡在二甲苯中使其完全脱水。在组织灌注石蜡后的短时间后,把它们放入(通常在一个小塑料盒内)65%石蜡浴,然后再放入一个模具中,最后凝固成小块,可经切片机切片。石蜡包埋法因其简单、成本低、样品保存持久、可行性高,因此很受欢迎。但石蜡包埋法也有缺点,即无法切的很薄(小于5微米)。为了能在高分辨率的光学显微镜和电子显微镜下观察切片,选择塑料包埋更好,因为可以切出很薄的组织切片,而且有助于保留组织形态。此外,塑料还可以成功包埋非常坚硬的组织(如骨)并切片。然而塑料包埋并不常用,因其比石蜡更昂贵,且会干扰组织染色。先前已冰冻的组织可以用最佳切片温度复合物(OCT)包埋,这是一种水溶性聚乙二醇树脂溶液。用OCT包埋残留少,从而背景信号低,所以是冷冻组织免疫组化染色的理想选择。OCT包埋的一个明显缺点就是在冰冻时变得不透明,在冰冻组织切片时定位较难。
| 方法 | 适用范围 | 利与弊 |
|---|---|---|
| 石蜡 | 甲醛固定组织 |
|
| 塑料 | 较硬的固定组织或需要切的很薄的组织样本(如样本需要电子显微镜观察时) |
|
| 最佳切片温度介质(OCT) | 快速冰冻组织 |
|
尽管使用福尔马林固定有许多优点,但是它却破坏了抗原表位的三维结构。可通过热诱导表位回复(HIER)逆转组织切片上的表位破坏。表位回复一般联合采用加热和酸/碱溶液;传统方法常把切片放在pH=6的柠檬酸钠缓冲溶液中加热,但有时高pH的缓冲液对某些抗原表位的回复更有效。切片和缓冲液可放在非常热的水浴、高压锅、高压锅或微波中加热,这取决于可用的设备。
切片标本可用好几种方法进行染色。通过酶标抗体和比色底物进行比色法是常用的。此法相对简单,反应稳定,切片可以通过标准显微镜读片分析。但是内源酶活性或链霉亲和素试剂对内源性生物素的非特异性结合,都可以提高背景信号强度,一般一个样品只能检测1-2个靶抗原。在这点考虑,通过免疫荧光或荧光染料标记的抗体检测标本更有优势,因为可以同时标记和检测多种靶抗原。但必须小心的是,偶联的荧光染料须避,以防荧光淬灭,因为这是不可逆的。
免疫金组织化学法可用作高分辨率分析。检测样品与偶联有不同大小的金颗粒的抗体孵育,从而可在单个样品中检测不同抗原。这些颗粒可以被高灵敏度和高分辨率的电子显微镜扫描检测到,从而可以在细胞和组织内对靶抗原进行非常精确的的定位。这种染色常用于验证亚细胞定位或特定的细胞衍生的结构如外泌体。
| 方法 | 适用范围 | 利与弊 |
|---|---|---|
| 酶联免疫比色法 | 标准光学显微镜 |
|
| 酶联免疫荧光法 | 荧光/共聚焦显微镜 |
|
| 金标记法 | 电子显微镜 |
|
检测和定性两种蛋白质之间的相互作用总是很有挑战性的。传统的方法,如双杂交试验,复杂而且呈现一定的局限性。邻位连接测试(PLA)是一种快速,灵敏且容易的技术可以同时检测和定量蛋白质相互作用。此外,它作为一种原位方法,有可能确定相互作用在贴壁细胞系和/或冷冻或石蜡包埋组织中的细胞内位置。 PLA也可以定量和检测在天然条件下单个内源蛋白。PLA于2002年首次被弗雷迪克森等人描述。 [25]. 自那时以来,这种技术已成为在不干扰的条件下分析天然蛋白质的相互作用,使用最广泛的方法之一。PLA方法的基础是使用寡核苷酸标记的两种抗体:这些抗体可以结合同一蛋白质的不同表位或两个不同蛋白质。当抗体接近彼此(约20-40纳米距离),例如,当两个待分析蛋白质在相互作用时,抗体上的寡核苷酸探针将与另外两个“接头寡核苷酸”杂交并结合,形成一个连续的环状DNA结构。这时,DNA聚合酶将放大该环状结构,环状结构仍然共价地跟一个PLA抗体连接。扩增的DNA分子可以使用标准的荧光方法来检测,从而直接显示两个待分析蛋白质之间的相互作用。PLA方法应用广泛:最常用的是在临床环境中确认生物标志物 [26-28] 。 PLA也已用于研究特定蛋白质在生物过程,如癌症的发展中的作用。近日,PLA已得到改进,直接检测翻译后修饰如乙酰化,磷酸化等。
这是在生物医学研究和临床中抗体最常见的应用方法,抗体可直接标记在细胞表面,并通过流式细胞仪直接检测各种细胞群。对单细胞悬液直接进行表面荧光抗体染色非常快捷;此染色方法不仅特异性好,而且灵敏度也高。此外,大多流式细胞仪能同时检测3个或更多的荧光抗体,因此可以检测到分化更细的细胞亚群及其分化、激活、凋亡相关的表面蛋白。抗体必须经过严格选择,确保抗体所针对的靶抗原表位表达在细胞表面,以及阻断剂(来源宿主的总免疫球蛋白IgG)应避免与某些细胞(如各种类型的淋巴细胞)上的Fc受体发生非特异性结合的(图9)。
此外,流式细胞术可用于检测细胞核,细胞质和核内体的蛋白,如转录因子和细胞因子。做此类检测时,还必须用甲醛和温和的离子去污剂(如皂苷素)对细胞进行固定、破膜,进行可逆地穿孔。表面染色应在细胞固定和破膜之前,以避免表面蛋白的抗原表位被破坏。
有激动作用的特异性抗体可作用于细胞表面受体以激活免疫细胞,此类抗体在体外也可结合和交联受体,从而激活细胞内的信号转导通路。例如,体外刺激T细胞活化可联合使用抗CD3抗体,CD3与T细胞受体(TCR)是密切相关的,CD3须激活后才能促进TCR和共刺激受体CD28的信号转导。然而,可以特异与一抗Fc段结合的二抗,必须完全交联受体从而激活细胞。另外,刺激性抗体也可在加入细胞培养前预先包被到培养板中(图6,9)。
抗体还可在体外用来封闭细胞表面的受体或中和可溶性因子。例如,在体外研究辅助性T细胞时,可在培养体系中加入特定的细胞因子从而促进T细胞分化为特定的辅助性T细胞亚群;而加入相关细胞因子的中和抗体时,可抑制T细胞分化为不同的细胞亚群。
荧光辅助细胞分选(FACS)是采用荧光标记的抗体结合特异性细胞,然后在特定荧光参数的基础上分选出的单细胞。此方法既快捷且特异性高;但是需要特定的流式细胞仪。偶联有磁珠的抗体也可用于分离或分选细胞,此过程称为磁性辅助细胞分选(MACS)。在MACS过程中,细胞被标记上带有特定标签的细胞表面标记的抗体。标记后的细胞随后与小磁珠孵育,小磁珠会结合到抗体的标签上。最后通过一个强有力的磁铁,可以非常容易地把标记有磁性微珠的细胞从未标记的细胞中分离出来(图9)。
为了研究某特定细胞群的功能,可以体内给予抗体以去除此类细胞。例如在免疫学研究中,可以通过抗体在小鼠体内去除特定的效应淋巴细胞亚群,从而可以观察机体对某种特定抗原的免疫应答变化。同样和上述体外研究类似,抗体可在体内封闭细胞的表面受体或中和可溶性因子。应用于此类方法的抗体,一般都是通过杂交瘤细胞大批量生产,以避免对异种抗原的反应,而且需要纯化以去除细胞培养试剂和其他可能的污染 [29] 。
总之,抗体是生物医学研究的宝贵工具,因其具有较高的敏感性和特异性,且相对容易生产,使用方法也灵活多样。已有的抗体应用方法继续在科学研究中发挥作用,以抗体为基础的新检测技术有望进一步发展,从而可进一步提升基础研究及转化研究实验室的科学检测分析能力。
质谱(MS)是一种分析技术,测量产物离子的质量 - 电荷(M / Z)比例,以检测,鉴别和量化在简单和复杂基质中的分子。如今,MS是在广泛的领域如蛋白质组学,药物开发,环境分析,生物医学研究中一个不可替代的工具。特别是蛋白质分析,因为使用了MS仪器,经历了非常迅速的发展,因此可以更准确和深入地定性类似的分子。然而,MS还有一些问题待解决,如生成的数据的数量巨大,还有高丰度蛋白质的存在掩盖感兴趣的蛋白质。后一点已被MS针对性方法部分克服,如多/选择反应监测(MRM/ SRM)。换句话说,MS灵敏度有一些问题,但也产生高度准确和具体的数据。在此框架下,MS与基于抗体的亲和力方法相结合,目标分子先用基于免疫的方法捕获,然后用MS分析,这个策略同时增加特异性和灵敏度。免疫测定和MS的结合开发了几个定量和定性的方法。最常用的简要报道如下。
MS技术允许复杂样品如血浆中的特定蛋白质的的绝对定量。这可以通过使用合成肽和重同位素的混合物(Absolute QUAntification, AQUA)来实现。在2004年Anderson等开发了稳定同位素标准和由抗肽抗体捕获(SISCAPA)方法,目标肽可通过使用固定在纳米柱的 [30] 抗肽抗体富集。该实验由4个步骤组成:ⅰ)用胰蛋白酶消化蛋白样品; 二)加入重同位素标准肽如AQUA; ⅲ)使用特异性抗肽抗体免疫富集iv)通过电喷雾电离质谱(ESI-MS)对肽绝对定量。SISCAPA有若干应用,例如用于乳腺癌患者的人血清转铁蛋白受体(sTfR)生物标志物的确定和定量 [31] 。然而,目标MS测定的改善使免疫-MS的研究倾向SISCAPA和MRM的结合,产生所谓的免疫MRM。
多反应监测质谱(MRM-MS)是具有高特异性和精度的有针对性的定量MS方法。为了增加该测定的灵敏度,有可能通过免疫亲和富集目标肽的混合物,由此进行免疫MRM。该技术是可重复的,多重的,并具有高敏感性和特异性。广泛延长使用免疫MRM的主要问题是缺乏经过验证的特异于该技术的抗体。抗体通常产生用于经典免疫市场(例如酶联免疫吸附,蛋白质印迹),而对于免疫MRM抗体应该对应短的直链的肽段。几项研究已经完成,调查在免疫MRM中单克隆抗体的使用。显然,这些产生的抗体优于多克隆抗体。不幸的是,单克隆抗体价格昂贵而且它们由杂交系统生产的时间长。最近,通过使用重组B细胞克隆的方法产生免疫MRM单克隆抗体抗胰蛋白酶肽抗原的可行性已被证明 [32] 。
免疫-MALDI(iMALDI)类似于SISCAPA方法:即使是在这种情况下,使用抗肽抗体捕获肽,但随后富集的样品点样到一个特定的目标并通过基质辅助激光解吸电离(MALDI)MS分析。这种方法已应用于诊断不同的病状,如高血压 [33] 。然而,iMALDI的主要限制之一在于无法多重测定。
质谱免疫测定(MSIA),于1995年 [34] 提出,蛋白质样品用包被有特异性抗体的珠子温育,然后洗脱。所获得的样品在全蛋白 [35] “自上而下”的方法之后,可以直接用MALDI-TOF MS分析。MSIA方法还可以结合目标MS方法,如SRM或MRM,洗脱样品需经过胰蛋白酶消化和随后的MS分析。最近由高塞尔等人报道这种方法可以自动和多重使用 [36] 。
- Sano T, Smith C, Cantor C. Immuno-PCR: very sensitive antigen detection by means of specific antibody-DNA conjugates. Science. 1992;258:120-2 pubmed
- Niemeyer C, Adler M, Wacker R. Detecting antigens by quantitative immuno-PCR. Nat Protoc. 2007;2:1918-30 pubmed
- O'Neill L, Turner B. Immunoprecipitation of native chromatin: NChIP. Methods. 2003;31:76-82 pubmed
- Ule J, Jensen K, Ruggiu M, Mele A, Ule A, Darnell R. CLIP identifies Nova-regulated RNA networks in the brain. Science. 2003;302:1212-5 pubmed
- Ule J, Jensen K, Mele A, Darnell R. CLIP: a method for identifying protein-RNA interaction sites in living cells. Methods. 2005;37:376-86 pubmed
- 17.
- Selth L, Gilbert C, Svejstrup J. RNA Immunoprecipitation to Determine RNA-Protein Associations In Vivo. CSH Protoc. 2009;2009:pdb.prot5234 pubmed
- Keene J, Komisarow J, Friedersdorf M. RIP-Chip: the isolation and identification of mRNAs, microRNAs and protein components of ribonucleoprotein complexes from cell extracts. Nat Protoc. 2006;1:302-7 pubmed
- Hellman L, Fried M. Electrophoretic mobility shift assay (EMSA) for detecting protein-nucleic acid interactions. Nat Protoc. 2007;2:1849-61 pubmed
- Egorina E, Sovershaev M, Østerud B. In-cell Western assay: a new approach to visualize tissue factor in human monocytes. J Thromb Haemost. 2006;4:614-20 pubmed
- Smith P, Wiltshire M, Davies S, Patterson L, Hoy T. A novel cell permeant and far red-fluorescing DNA probe, DRAQ5, for blood cell discrimination by flow cytometry. J Immunol Methods. 1999;229:131-9 pubmed
- Smith P, Blunt N, Wiltshire M, Hoy T, Teesdale-Spittle P, Craven M, et al. Characteristics of a novel deep red/infrared fluorescent cell-permeant DNA probe, DRAQ5, in intact human cells analyzed by flow cytometry, confocal and multiphoton microscopy. Cytometry. 2000;40:280-91 pubmed
- Fredriksson S, Gullberg M, Jarvius J, Olsson C, Pietras K, Gústafsdóttir S, et al. Protein detection using proximity-dependent DNA ligation assays. Nat Biotechnol. 2002;20:473-7 pubmed
- Seaman W, Wofsy D. Selective manipulation of the immune response in vivo by monoclonal antibodies. Annu Rev Med. 1988;39:231-41 pubmed
- Anderson N, Anderson N, Haines L, Hardie D, Olafson R, Pearson T. Mass spectrometric quantitation of peptides and proteins using Stable Isotope Standards and Capture by Anti-Peptide Antibodies (SISCAPA). J Proteome Res. 2004;3:235-44 pubmed
- Nelson R, Krone J, Bieber A, Williams P. Mass spectrometric immunoassay. Anal Chem. 1995;67:1153-8 pubmed
- Gauthier M, Pérusse J, Awan Z, Bouchard A, Tessier S, Champagne J, et al. A semi-automated mass spectrometric immunoassay coupled to selected reaction monitoring (MSIA-SRM) reveals novel relationships between circulating PCSK9 and metabolic phenotypes in patient cohorts. Methods. 2015;81:66-73 pubmed 出版商
- 来邦网
- 英文来邦