An updated review of PCR methods, including conventional, real-time, microfluidic, and drop digital PCR.
聚合酶链反应(PCR)是一种很普及的技术,广泛应用于临床诊断和分子生物学研究。 PCR是由DNA聚合酶在体外扩增特定的核酸(NA)序列。PCR技术的飞跃发生在1983年,当Kary Mullis推广使用了伴随温度循环的热稳定聚合酶 [1-3] 。PCR的普遍应用在于其能把数量很少的靶核酸序列,扩增成大量的PCR产物,然后通过下游方法检测,例如通过琼脂糖凝胶电泳观察核酸(NA)产物。这是由于核酸(NA)序列的指数扩增,使得起始模板(靶核酸序列)扩增产生数百万的拷贝。
PCR反应扩增靶核酸序列是通过使用DNA聚合酶,引物和核苷酸。 PCR反应的模板可能是任何靶核酸序列,核酸的来源可能是DNA,RNA或cDNA。引物是体外合成的核酸短序列。引物的设计是互补结合于特定靶核酸模板的反义链,引物的长度通常是15-40个碱基。引物最好是缺少二级结构并且不彼此互补,以防止引物二聚体的形成。多种DNA聚合酶都可用于PCR,但最广泛使用的是耐热的Taq DNA聚合酶。这种酶根据模板的核酸序列进行引物延伸(在引物末端添加dNTPs或核苷酸)。
PCR反应通常是20-40个温度循环。核酸模板序列的变性是在95℃进行的。引物退火结合与靶序列是在温度冷却至37-60℃进行的。引物的核苷酸延伸是DNA聚合酶在60-72℃的范围内进行的。常规的循环条件是起初95℃持续5分钟,使得所有的核酸模板变性,接着是重复2-40个温度循环(95℃持续30秒,60℃持续30秒,72℃持续1分钟)。对于具体试验,在每个温度上持续的时间可以优化。例如,很短的靶序列的扩增相比于很长的靶序列,在每个温度上需要持续的时间就要短得多。每个温度循环获得的靶序列产物都是前一个循环的产物的2倍。这就导致了原始模板的指数扩增,通常获得的产物是原始靶核酸序列的数百万或数十亿的拷贝。
基本PCR反应有三个时期。指数时期是每个温度循环扩增的核酸产物都是确切倍增。实时PCR检测就是在指数时期进行的。线性时期发生的反应减慢是由于试剂的消耗和产物的降解。最后时期是平台期,就是反应停止,不再生成扩增产物了。常规PCR反应就是在平台期通过凝胶电泳分析PCR反应产物。
PCR产物的下游检测有许多种方法。一种常用的观察方法是通过琼脂糖凝胶电泳。这包括通过电泳分离核酸片段,使用嵌入染料如溴化乙锭或SybrSafe染色核酸片段,随后使用紫外线光源和成像系统检测(图2)。

实时PCR使用专门的热循环仪检测每个反应孔的荧光信号。这个信号指示的是反应管或反应孔所含的双链核酸的量。这个信号表示为相对荧光单位,是热循环仪软件相对于温度循环周期数而绘制的(图3)。
PCR广泛应用于科研,临床和法医领域。在分子生物学研究中,PCR常用于基因工程,DNA测序,基因表达分析。 在临床实验室中,PCR对感染性疾病的检测是至关重要的。在司法鉴定中,PCR的应用包括亲子鉴定和DNA指纹。这些应用都是利用了PCR极高的敏感性,即使是来自人的一根头发中靶核酸序列,PCR也能检测出来。表一列出了PCR的主要应用及其参考文献。
| 应用 | 参考文献 |
|---|---|
| 食物中病原体的检测 | [4, 5] |
| 感染性疾病的诊断 | [6] |
| 基因分型 | [7, 8] |
| 人类遗传突变的检测 | [9, 10] |
| microRNA的表达谱 | [11, 12] |
| 基因表达分析 | [13-15] |
| DNA测序 | [16, 17] |
| 法医 - 遗传指纹 | [18-20] |
| 法医- 亲子鉴定 | [21] |
| 基因工程 | [22, 23] |
PCR方法有许多种,每种方法都有各自的优点和局限性。标准或常规PCR是PCR反应的最基本类型。能给出定性结果,并且需要PCR结束后的DNA检测或观察步骤。常规PCR的主要优点是成本相当低,并且几乎所有的实验室都有可用的常规PCR仪。通常情况下,常规PCR反应结束后,通过琼脂糖凝胶电泳按照核酸片段的大小分离产物。 使用溴化乙锭或Sybr Safe等嵌入染料和紫外线光源观察DNA扩增产物。 PCR反应特异性的初步确定是通过产物的片段大小相比于DNA梯状条带,DNA梯状条带是已知大小的DNA片段的混合物。产物的电泳条带可从凝胶中切割分离,条带中的DNA通过纯化和测序,从而更可靠地确定PCR反应特异性。图2是琼脂糖凝胶电泳图的一个示例,左侧是DNA梯状条带,右侧是几个PCR反应产物的电泳。标准或常规PCR的局限性是灵敏度低,并且不能给出定量结果。
| 实验需求 | PCR 方法 | 优点 | 局限性 |
|---|---|---|---|
| 确定在几个样品中是否存在靶核酸序列 | 标准或常规 |
|
|
| 在许多样品中靶核酸序列的定量检测 | 实时 |
|
|
| 测定几个样品的多个靶核酸序列的定量结果 | PCR 芯片 |
|
|
| 野外实地检测靶核酸 | 微流控芯片 |
|
|
| 检测极低丰度的靶核酸或者需要极为精确的定量 | 数字和液滴的数字 |
|
|
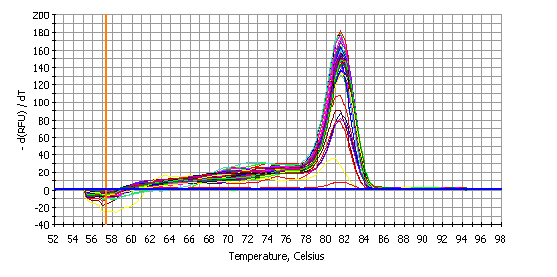
实时PCR是最近的,但已经非常普遍的PCR方法,与常规PCR相比,实时PCR有几个优点。首先,可以实时检测PCR产物,所以就不需要在PCR结束后通过琼脂糖凝胶电泳观察检测DNA了。此外,实时PCR是定量的和特异的。通过与已知量的DNA的标准曲线相比,可以确定样本的靶序列起始量。PCR反应之后,通过使用特定的核酸探针和/或熔解曲线分析,使其特异性增加。参见图4示例的单一的PCR产物的熔解曲线。单峰的存在表明PCR反应得到了单一的扩增产物。多峰的存在表明PCR反应得到了多个扩增产物,由此PCR检测的反应体系需要进一步优化和改进。实时PCR的局限性是成本增加,并且需要专门的热循环仪,而且靶核酸序列起始量的定量精确度有限。
PCR芯片是使用实时PCR热循环仪,基于实时PCR的SYBR green检测。 PCR芯片是在96孔板各孔中加入不同的基因特异引物,可以检测同一样品中多达88个基因的表达,以及8个标准或对照反应。PCR芯片在96孔板的每一个孔中检测单一基因的特定产物。PCR芯片通常包括对照反应,能给出基因表达的定量结果。PCR芯片通常是用于一个特定的生物学通路(例如,DNA修复,细胞周期,或氧化应激)的一组相关基因的表达分析。PCR芯片的优点是可以同时检测一个样品中多个不同基因的表达。局限是每个样品都需要分别处理,所以非常耗时,而且也很昂贵。
微流控芯片PCR是使用微细加工和微流体,与常规或实时PCR相比,微流控芯片PCR可以更快速地扩增DNA。此外,微流控芯片PCR的体积小,并且集成了检测元件,所以具有更好的便携性和便于野外使用。数字和液滴的数字PCR能够分隔核酸分子,不需要标准曲线就能定量PCR终产物。这样就可以非常精确地测定拷贝数,并且能够检测到非常低拷贝数的目标核酸序列。虽然微流控芯片PCR是强大和精确的,但是许多研究者都用不起。这是由于所需的微加工设备,或需要购买的预制芯片,是相当昂贵的。然而,微流控芯片PCR发展迅速,成本可能很快就会显著降低。
生物学研究还使用了许多其他PCR方法。基因分型通常使用等位基因特异的PCR [24] 。表观遗传学研究是主要使用甲基化特异的PCR。这种技术检测基因组DNA中CpG岛的甲基化 [25] 。降落PCR(Touchdown PCR)是通过随着反应的进行,逐步降低退火温度,从而减少非特异性扩增。这种技术有利于特定靶序列扩增产量的提高 [26] 。
虽然PCR是一种很普及的技术,但是仍有很多机会可以改进,从而缩短反应所需的时间和降低反应液的体积。反应速度取决于加热和温度循环机制的类型,以及反应液的体积,因此是PCR改进的首要目标。基于金属块加热的PCR系统,使用间接传导加热,是很普及的,但是金属块的热质量很高,并且需要的反应液体积比较大,所以热变化率相当缓慢(3ºC/s) [27] 。Roche Lightcycler使用对流加热(convective heating),并且需要的反应液体积只有几微升,所以温度变化的速度很快(10ºC/s) [28] 。 微芯片PCR设备进一步加快了反应的速度和降低了反应液的体积。这些芯片最常用的导热原材料是硅或玻璃。这些芯片是热电加热,通过基于对流加热的旋转平台,或嵌入式电阻加热器加热 [29-31] ,并且需要的反应液体积只有几微升。微芯片PCR还可以使用钨丝灯的红外线辐射直接加热 [32] 。也可以使用激光器的红外导直接加热 [33, 34] 。这两种方法都能在几分钟之内得到PCR结果,并且需要的反应液体积只有几皮升(picoliters)。
假阳性和假阴性PCR结果都影响PCR检测的有效性,所以就有必要优化PCR反应,以防止这些假象。假阳性结果是指即使在反应体系中没有加入起始模板,也能检测到DNA扩增产物。这可能是由于存在污染。必须在洁净实验室操作,以避免气雾化的DNA不经意地污染反应体系。假阴性结果是指虽然靶核酸是存在的,但是却无法检测到扩增产物。原因可能是引物的问题,或者是温度循环参数不是最优化的,或者是扩增的产物量低于系统的检测下限。为了减少假阴性的发生,就有必要对PCR检测的体系设计和温度循环条件进行优化。这包括选择高质量的引物 [35-37] ,循环过程中温度的优化,使用高质量的核酸模板。
与常规PCR相比,实时PCR有显著的优点。实时PCR使用检测系统测量的荧光指示,通常是96孔板的封闭管中的DNA扩增产物。实时PCR不需要在PCR结束之后通过琼脂糖凝胶电泳检测,从而显著缩短了获得结果所需的时间。 在每个温度循环后,检测PCR产物,这样就可以测量PCR的反应动力学。与通过琼脂糖凝胶电泳检测DNA相比,PCR产物的荧光检测也更加敏感,所以靶DNA的两倍量差异,实时PCR就能够检测出来,但是常规PCR和琼脂糖凝胶就不可能检测出来。此外,使用基于探针的实时检测,PCR扩增产物的检测就是序列特异的。
DNA结合染料: 实时PCR采用与PCR产物相互作用的荧光染料或探针。荧光检测的两种主要类型是DNA结合染料,例如SYBR Green,或荧光标记的序列特异探针,例如TaqMan或分子信标探针(Molecular Beacon probes)。当DNA结合染料结合于任何双链DNA片段时,染料就发出荧光信号 [38] 。当只有单链核酸存在时,这些染料就不结合于核酸,发出的荧光信号就很弱。虽然SYBR Green是最常用的,但是其他一些DNA结合染料也有使用。这些染料包括SYTO 9, [39] SYTO-13, SYTO-82, [40] 和 EvaGreen [41] 。因为这些染料的荧光信号的检测不是序列特异的,所以就必须进行熔解温度分析,以确保PCR产物是单一的产物 [42] 。参见图4示例的单一的PCR产物的熔解曲线。如果熔解曲线有多个峰,那么就表明存在多个PCR产物,PCR检测体系就需要进一步优化。虽然这种方法可能是耗时的,但是SYBR Green和其他DNA结合染料仍然很受欢迎,这是因为基于染料的检测方法在价格成本上明显低于基于探针的检测方法。
核酸酶依赖的探针: 实时PCR采用的检测化学的第二种主要类型是序列特异性荧光标记的探针。这些探针系统可以是核酸酶依赖的探针或者是简单的杂交探针。核酸酶切割的探针包括TaqMan, HybProbe(两种寡核苷酸),小沟结合(MGB)探针,锁核酸(LNA)探针。这些探针与PCR扩增产物中的目标核酸序列互补结合,探针的两端分别共价连接一个报告荧光染料和一个淬灭荧光染料。这些系统使用的是萤光共振能量转移(FRET)技术。当染料彼此接近的时候,报告染料的荧光信号被淬灭,因此就检测不到任何信号了。然而,当染料彼此分开的时候,报告染料的荧光不被淬灭,因此就能检测到信号 [43] 。探针退火结合的序列是在PCR引物的结合位点中,Taq DNA聚合酶在延伸引物产生PCR产物的同时,其5'外切酶活性把探针的末端切除。淬灭染料被切除之后,报告染料就能在激发下,产生荧光信号。循环探测技术(CPT)的探针包含RNA核苷酸,所以不同于TaqMan类型的探针。CPT探针杂交于靶序列之后形成RNA-DNA双链。 然后用RNaseH酶把探针的淬灭染料切除 [44] 。
杂交探针: 虽然杂交探针也是序列特异的,但是不需要核酸外切酶把杂交探针的染料切除。杂交探针包括分子信标(Molecular Beacons),在两个反向重复序列之间有一个单环区域,形成了一个发夹结构。探针变性,然后退火结合到目标序列,发夹就被打开了,荧光染料彼此分开,足以使得报告染料生成增强的荧光信号 [45] 。其他检测探针技术,包括Scorpion(探针和引物合并在一个分子上 [46] ),LUX (Light Upon eXtension)检测 [47] 。 Lux 引物的发夹结构淬灭了 Lux 引物探针3'端的荧光染料信号。一旦引物结合到靶序列,随着DNA聚合酶延伸引物序列,染料的荧光信号就增强了 [47] 。
实时PCR热循环仪系统检测的荧光强度,与PCR扩增产物的积累是成正比的。然而,结果分析只限于实时PCR反应指数时期的数据,因为这是精确定量的最佳数据点。阈值循环数(CT)是实时PCR反应管中的荧光强度超过阈值时温度循环的周期数,荧光强度的阈值是设定在基线以上,但是在扩增的指数时期之内。实时PCR一般采用两种定量测定方法。相对定量是把试验处理的样本与对照的样本相比较,比照标准化的内参基因表达量,确定靶序列基因表达量的相对比例。关键是试验处理方法不会对使用的内参基因的表达量造成影响。相对定量的主要优点是因为不需要建立校准曲线,所以缩短了检测时间。常用的方法包括相对标准曲线法,以及比较CT法(ΔΔCT)。后者的数学模型计算参见 Pfaffl [48] 。
绝对定量是通过把样品的扩增信号对比于靶DNA的标准曲线,检测样品中靶序列的实际的起始量。校准曲线可以是基于核酸分子(可以是重组质粒DNA其中插入了亚克隆扩增序列,实时PCR产物,或合成的大片段寡核苷酸)的稀释,。质粒DNA被广泛使用,是因为它的稳定性高于PCR产物或寡核苷酸。然而,较短模板的准备所需的时间也较短。通过对已知浓度的质粒(插入了亚克隆扩增序列)进行实时PCR,得到质粒的标准曲线,画图关联CT值与靶序列起始拷贝数的对数(图五)。 CT值与起始拷贝数的对数是成反比的 [49] 。通过对标准曲线的线性回归,确定试验样品的靶序列起始拷贝数 [50] 。

自1990年以来,对PCR反应和热循环仪的微型化创新,体现在微流控芯片PCR。例如,在1998年,Northrup et. al报道了他们创建的一个微型分析热循环仪(MATCI),是硅材料的并且反应孔集成了加热器 [51] 。MATCI仪在PCR循环进行的同时实时检测荧光信号,仪器在便携性上等同于公文包。此后,微流控芯片PCR领域开始了腾飞,技术进步包括多种加工材料和构造 [52] 。
大多数PCR芯片的反应腔是硅材料的。硅具有优异的导热性,在温度循环过程中可以实现快速温度变化。然而,芯片材料也可以是玻璃,聚合物包括聚碳酸酯和聚二甲基硅氧烷,陶瓷,317不锈钢。芯片构造可以是基于静态固定反应腔,也可以是基于动态连续流动的。固定反应腔的芯片,PCR反应液是静态的,反应腔进行温度循环。这些芯片可能是单腔或多腔。动态连续流动的芯片,在PCR扩增的温度循环过程中,样品被泵驱动通过微通道。这些芯片的样品流速决定了温度转换的时间。 微芯片PCR缩短了检测时间,试剂消耗低,加热和冷却的温度循环速度快,并且降低了耗电量。 微芯片PCR可能需要下游处理检测DNA,例如琼脂糖凝胶电泳或毛细管电泳。然而,在芯片上也可能包括DNA检测方法,例如毛细管电泳,荧光检测或DNA杂交方法 [29, 53] 。 微芯片PCR技术还在继续发展,包括芯片检测系统的集成,以及加工材料和温度循环方法的优化。
基于微流控腔的数字PCR(cdPCR)方法无需使用标准曲线,就能够进行核酸的绝对定量 [54] 。数字cdPCR是样品被分隔成离散的油包水液滴,液滴中可能没有任何的靶核酸,或可能有一个或多个拷贝的靶核酸序列。 在PCR扩增之后,检测靶序列的存在,通过计数阳性液滴在液滴总数中所占的比例,进行浓度计算。 “液滴”数字PCR是最尖端的数字PCR方法。“液滴”数字PCR的液滴分隔数目是数字PCR的25倍,可以非常精确地估算靶序列的拷贝数 [55] 。
- Mullis K, Faloona F, Scharf S, Saiki R, Horn G, Erlich H. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb Symp Quant Biol. 1986;51:263-73 pubmed
- Mullis K, Faloona F. Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction. Methods Enzymol. 1987;155:335-50 pubmed
- Saiki R, Scharf S, Faloona F, Mullis K, Horn G, Erlich H, et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science. 1985;230:1350-4 pubmed
- Malorny B, Tassios P, Radstrom P, Cook N, Wagner M, Hoorfar J. Standardization of diagnostic PCR for the detection of foodborne pathogens. Int J Food Microbiol. 2003;83:39-48 pubmed
- McBride L, Koepf S, Gibbs R, Salser W, Mayrand P, Hunkapiller M, et al. Automated DNA sequencing methods involving polymerase chain reaction. Clin Chem. 1989;35:2196-201 pubmed
- Foster A, Laurin N. Development of a fast PCR protocol enabling rapid generation of AmpFℓSTR® Identifiler® profiles for genotyping of human DNA. Investig Genet. 2012;3:6 pubmed
- Decorte R, Cassiman J. Forensic medicine and the polymerase chain reaction technique. J Med Genet. 1993;30:625-33 pubmed
- Schlenk J, Seidl S, Braunschweiger G, Betz P, Lederer T. Development of a 13-locus PCR multiplex system for paternity testing. Int J Legal Med. 2004;118:55-61 pubmed
- Saiki R, Bugawan T, Horn G, Mullis K, Erlich H. Analysis of enzymatically amplified beta-globin and HLA-DQ alpha DNA with allele-specific oligonucleotide probes. Nature. 1986;324:163-6 pubmed
- Herman J, Graff J, Myöhänen S, Nelkin B, Baylin S. Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands. Proc Natl Acad Sci U S A. 1996;93:9821-6 pubmed
- Don R, Cox P, Wainwright B, Baker K, Mattick J. 'Touchdown' PCR to circumvent spurious priming during gene amplification. Nucleic Acids Res. 1991;19:4008 pubmed
- Wittwer C, Ririe K, Andrew R, David D, Gundry R, Balis U. The LightCycler: a microvolume multisample fluorimeter with rapid temperature control. Biotechniques. 1997;22:176-81 pubmed
- Khandurina J, McKnight T, Jacobson S, Waters L, Foote R, Ramsey J. Integrated system for rapid PCR-based DNA analysis in microfluidic devices. Anal Chem. 2000;72:2995-3000 pubmed
- Liao C, Lee G, Liu H, Hsieh T, Luo C. Miniature RT-PCR system for diagnosis of RNA-based viruses. Nucleic Acids Res. 2005;33:e156 pubmed
- Huhmer A, Landers J. Noncontact infrared-mediated thermocycling for effective polymerase chain reaction amplification of DNA in nanoliter volumes. Anal Chem. 2000;72:5507-12 pubmed
- Rozen S, Skaletsky H. Primer3 on the WWW for general users and for biologist programmers. Methods Mol Biol. 2000;132:365-86 pubmed
- Schneeberger C, Speiser P, Kury F, Zeillinger R. Quantitative detection of reverse transcriptase-PCR products by means of a novel and sensitive DNA stain. PCR Methods Appl. 1995;4:234-8 pubmed
- Monis P, Giglio S, Saint C. Comparison of SYTO9 and SYBR Green I for real-time polymerase chain reaction and investigation of the effect of dye concentration on amplification and DNA melting curve analysis. Anal Biochem. 2005;340:24-34 pubmed
- Gudnason H, Dufva M, Bang D, Wolff A. Comparison of multiple DNA dyes for real-time PCR: effects of dye concentration and sequence composition on DNA amplification and melting temperature. Nucleic Acids Res. 2007;35:e127 pubmed
- Eischeid A. SYTO dyes and EvaGreen outperform SYBR Green in real-time PCR. BMC Res Notes. 2011;4:263 pubmed
- Ririe K, Rasmussen R, Wittwer C. Product differentiation by analysis of DNA melting curves during the polymerase chain reaction. Anal Biochem. 1997;245:154-60 pubmed
- Holland P, Abramson R, Watson R, Gelfand D. Detection of specific polymerase chain reaction product by utilizing the 5'----3' exonuclease activity of Thermus aquaticus DNA polymerase. Proc Natl Acad Sci U S A. 1991;88:7276-80 pubmed
- Duck P, Alvarado-Urbina G, Burdick B, Collier B. Probe amplifier system based on chimeric cycling oligonucleotides. Biotechniques. 1990;9:142-8 pubmed
- Tyagi S, Kramer F. Molecular beacons: probes that fluoresce upon hybridization. Nat Biotechnol. 1996;14:303-8 pubmed
- Whitcombe D, Theaker J, Guy S, Brown T, Little S. Detection of PCR products using self-probing amplicons and fluorescence. Nat Biotechnol. 1999;17:804-7 pubmed
- Nazarenko I, Lowe B, Darfler M, Ikonomi P, Schuster D, Rashtchian A. Multiplex quantitative PCR using self-quenched primers labeled with a single fluorophore. Nucleic Acids Res. 2002;30:e37 pubmed
- Pfaffl M. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Res. 2001;29:e45 pubmed
- Higuchi R, Fockler C, Dollinger G, Watson R. Kinetic PCR analysis: real-time monitoring of DNA amplification reactions. Biotechnology (N Y). 1993;11:1026-30 pubmed
- Bustin S. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. J Mol Endocrinol. 2000;25:169-93 pubmed
- Northrup M, Benett B, Hadley D, Landre P, Lehew S, Richards J, et al. A miniature analytical instrument for nucleic acids based on micromachined silicon reaction chambers. Anal Chem. 1998;70:918-22 pubmed
- Zhang C, Xing D. Miniaturized PCR chips for nucleic acid amplification and analysis: latest advances and future trends. Nucleic Acids Res. 2007;35:4223-37 pubmed
- Vogelstein B, Kinzler K. Digital PCR. Proc Natl Acad Sci U S A. 1999;96:9236-41 pubmed