从培养的细胞中分离病毒,然后采用免疫荧光和分子生物学技术进行病毒核酸检测已被成功地用于病毒的鉴定。目前最常用的病毒定量检测方法有以下三大类:用于病毒感染力检测的技术,如病毒空斑形成试验,半数组织培养感染剂量TCID50测定和免疫荧光等;病毒核酸和病毒蛋白检测技术,如实时定量多聚酶链反应(qPCR),免疫印迹,免疫沉淀,酶联免疫吸附测定(ELISA)和血凝试验等;还有就是那些直接对病毒颗粒进行计数的方法,如流式细胞分析或透射电镜技术。由于以上每种方法均存在一些局限性(见表1),目前仍需要发展新的更为优化的病毒鉴定和定量手段以克服目前检测手段的缺点。本文主要对目前实验和诊断中最常用的几种病毒鉴定和定量方法进行综述。
| 方法 | 检测原理 | 重复性 | 时间 | 工作量 | 费用 |
|---|---|---|---|---|---|
| 病毒空斑试验 | 感染力检测 | 差 | 数天 | 大 | 低 |
| TCID50, LD50, EID50 | 感染力检测 | 差 | 数天 | 大 | 低 |
| 免疫荧光斑块试验 | 感染力检测 | 差 | 数天 | 大 | 高 |
| qPCR | 病毒核酸检测 | 很高 | 数小时 | 中等 | 高 |
| 免疫印迹法(immunoblotting) | 病毒蛋白检测 | 好 | 数天 | 中等 | 低 |
| 免疫沉淀法(immunoprecipitation) | 病毒蛋白检测 | 好 | 数天 | 中等 | 低 |
| ELISA | 病毒蛋白检测 | 好 | 数小时 | 中等 | 低 |
| 血凝试验(hemagglutination assay) | 病毒蛋白检测 | 好 | 数小时 | 中等 | 低 |
| 病毒流式细胞术(viral flow cytometry) | 病毒颗粒计数 | 很好 | 数小时 | 大 | 高 |
| 透射电镜法 | 病毒颗粒计数 | 很好 | 数周 | 大 | 高 |
在1954年, J. F. Enders 首次提出了基于细胞培养体系的动物病毒分类方法,根据感染细胞后产生的不同效应将病毒分为以下4类:致细胞变性的病毒,致包涵体形成和细胞变性的病毒、致细胞融合形成多核细胞的病毒、以及那些不导致细胞病变效应(CPE)的病毒 [1] 。自那以后,细胞培养成了病毒体外 分离检测的常规手段 [2] 。通过观察宿主细胞的形态变化可对致细胞病变病毒的类型进行一个初步的鉴定。病毒对宿主细胞常见的损害包括使细胞变圆,细胞质地发生改变(颗粒状或透明玻璃质),以及发生细胞融合(见 ASM Microbe Library)。 病毒感染导致的可见的细胞损伤程度因病毒的毒株,宿主细胞类型和感染复数(MOI,即病毒颗粒与细胞的比率)而异;细胞病变效应最快可在病毒感染后2天即可观察到,慢的要在4周才开始出现。虽然大部分实验室把传统的与先进的实验技术相结合来优化病毒诊断,从培养的细胞中分离病毒仍然是目前主要使用的方法,尤其是当需要分辨病毒是否有活力的时,当必需要分离活病毒时,以及当感染性并不是某个病毒的特征时 [2] 。传统的从培养细胞中分离病毒的方法被认为是病毒检测和诊断的金标准,这一技术通常被用来与其它检测技术进行比较 [3] 。
临床样本收集及病毒分离的方法在各实验室间可能存在很大差异,但一些常规的病毒样本收集、运输和病毒培养方法可参照几本参考书里的建议 [4] 。同样,细胞培养接种的方法也需要根据样本的种类(例如,痰液样本和粪便样本)和疑似的病毒毒株类型而定。为了加快病毒分离的过程,病毒快速培养方法如离心增强技术(Shell vial法)的出现使试验周期大大缩短,从1周缩短至1-2天 [5] 。近年来,Shell vial法显著提高了各种病毒包括流感病毒,登革热和呼吸道病毒等的分离效率 [6-8] 。病毒感染培养的单层细胞后,每天在显微镜下观察致细胞病变效应(CPE),病毒包涵体形成情况,细胞融合情况,以及细胞空泡形成情况。
- 细胞生长培养基:含10% FBS(胎牛血清), 4-6 mM Glutamine(谷氨酰胺)的高葡萄糖DMEM,若有需要可加入青霉素,链霉素,庆大霉素,两性霉素等
- 维持培养基:上述新鲜的细胞培养基,但血清FBS浓度减少至为2%
- 宿主细胞:铺满单层细胞的8孔培养腔室玻片(chamber slides)
- PBS磷酸缓冲液
- Bouin's固定液
- 吉姆萨(Giemsa)缓冲液
- 吉姆萨(Giemsa)染色液
- 丙酮,丙酮:二甲苯(2:1),丙酮:二甲苯(1:2),二甲苯
- 中性树胶
- 移液枪头 (10 到100 微升)
- 移液管
- 生物安全柜(超净台)
- 二氧化碳培养箱
- 倒置显微镜
注: Labome有一篇综述对细胞培养基的各种组分进行了详细描述。 Labome.
- 接种宿主细胞至Chamber Slides:选择合适的细胞接种密度(比如8-孔Chamber Slides,接种30,000细胞/孔),培养基为细胞生长培养基(10%FBS-DMEM)。轻轻地前后左右摇晃chamber slides使细胞分布均匀。将细胞置培养箱培养过夜,第二天,显微镜下观察细胞确认细胞是否分布均匀并达到80%以上的融合度。
- 制备不同稀释度的病毒液:标记6支无菌离心管,第1管加入990 μl细胞生长培养基,剩下5管加入900μl细胞生长培养基。按以下方法进行梯度稀释:在第1管中加入10 μl病毒原液(稀释度1:100)充分混匀,然后从第1管吸100 μl至第2管中混匀,依次类推,进行10倍梯度稀释。管1至管6的稀释度分别为 10-3 到 10-7。
- 感染细胞:吸去培养孔中的培养液,每孔加入0.5 mL维持培养液(2%FBS-DMEM),再加入100 μl不同稀释度(10-2 至 10-7稀释)的病毒液至其中一孔,留一孔不加作为空白对照。将细胞置二氧化碳培养箱37oC 或 34oC 培养1-4周,追踪观察致细胞病变效应(CPE)发生情况。
- 吉姆萨染色:一旦观察到细胞病变效应,轻轻地用PBS将细胞洗3次,每次5min,然后加入Bouin's固定液固定10 min。用吉姆萨缓冲液洗3次,然和加入Giemsa染液染色1 h,再用吉姆萨缓冲液清洗后依次用丙酮处理15s,2:1的丙酮-二甲苯处理30s,1:2的丙酮-二甲苯处理30s,最后用二甲苯处理10 min紧接着用中性树胶封片。显微镜下观察CPE,包涵体,细胞融合及病毒空泡形成情况。病毒感染细胞后形成的CPE图片范例可以在很多图谱,网站和博客上找到,例如ASM Microbe Library viral cytopathic effect。
免疫荧光技术可用于鉴定病毒抗原,目前被广泛应用于临床样本病毒感染的快速检测 [9-12] 。免疫荧光染色通常被认为是一种快速(1-2h)、敏感且比较特异的病毒鉴定手段 [9-12] 。遗憾的是,免疫荧光检测并不能鉴别所有的病毒毒株,比如肠道病毒,因为大部分肠道病毒的单克隆抗体敏感性差,跟鼻病毒发生交叉反应的情况非常普遍 [13] 。不过,免疫荧光检测已被成功地用于流感病毒感染性控制和病毒活力监测 [10, 11] 。根据疾控中心推荐,在流感病毒活动度低的时候,发生假阳性的可能性较高,阳性结果应当经间接免疫荧光、病毒培养,或RT-PCR方法证实;而在病毒活动高峰期的时候,必须用间接免疫荧光、病毒培养,或RT-PCR等检测方法确认,因为阴性结果并不能完全排除病毒感染的可能性 (参照 FDA文件)。 有趣的是,通常认为免疫荧光的敏感度比ELISA或PCR要低,然而最近有文献报道:对于水痘-带状疱疹病毒(VZV)的快速检测,间接免疫荧光法比传统的细胞培养法更优越 [14] 。对于儿童皮肤粘膜单纯疱疹病毒(HSV)的检测,间接免疫荧光法的准确度却很低(敏感度61%,特异性99%) [15] 。再者,单纯疱疹病毒的单克隆抗体CHA437虽然与水痘-带状疱疹病毒、巨细胞病毒、EB病毒等无交叉反应,但它在实际样本检测时只有84.6%敏感度和95.7%的特异性 [16] 。而对于严重急性呼吸道冠状病毒(SARS病毒),抗原检测试验从17份SARS病人样本的11份(65%)中检测到含有SARS冠状病毒 [17] 。
免疫荧光技术是一种公认的实验诊断方法,尽管有时候因为使用的抗体成本较高使得检测价格可能比较昂贵。另外,由于一些商品化抗体的特异性不够理想,发生非特异结合或交叉反应,也会导致结果出现一些误差。
- 细胞生长培养基:含10%FBS(胎牛血清), 4-6mM Glutamine(谷氨酰胺)的高葡萄糖DMEM,若有需要可加入青霉素,链霉素,庆大霉素,两性霉素等
- 宿主细胞:铺满单层细胞的8孔培养腔室玻片(chamber slides)
- PBS磷酸缓冲液
- 一抗
- FITC标记的二抗
- 细胞核染料DAPI
- 5-4 %多聚甲醛(PFA,pH7.4),或者甲醇
- 移液枪头 (10 到100 微升)
- 离心管
- 生物安全柜(超净台)
- 二氧化碳培养箱
- 荧光显微镜
- 接种宿主细胞至Chamber Slides:选择合适的细胞接种密度(比如8-孔Chamber Slides,接种30,000细胞/孔),培养基为细胞生长培养基(10%FBS-DMEM)。轻轻地前后左右摇晃chamber slides使细胞分布均匀。将细胞置培养箱培养过夜,第二天,显微镜下观察细胞确认细胞是否分布均匀并达到80%以上的融合度。
- 病毒感染:每孔细胞加0.1 mL病毒储存液,留一孔不加作为阴性对照。将细胞放回CO2 培养箱,37°C 或 34°C 培养48h。
- 免疫荧光染色观察:病毒感染48h后,吸去培养液,将细胞用预冷的甲醇固定5min(也可用新鲜制备的4%多聚甲醛固定)后,用含0.2% triton X的PBS室温破膜5min。将细胞用PBS清洗后,加入推荐浓度的一抗孵育2h,PBS清洗5遍,然后再加入荧光(Alexa fluor 488 或FITC)标记的二抗进行孵育,PBS清洗5遍,加入DAPI染细胞核,在相差及荧光显微镜下观察结果。
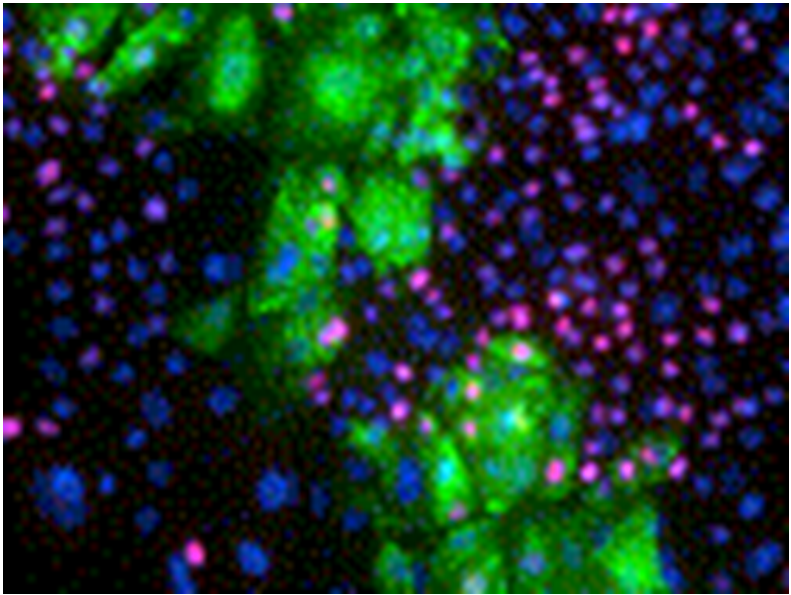
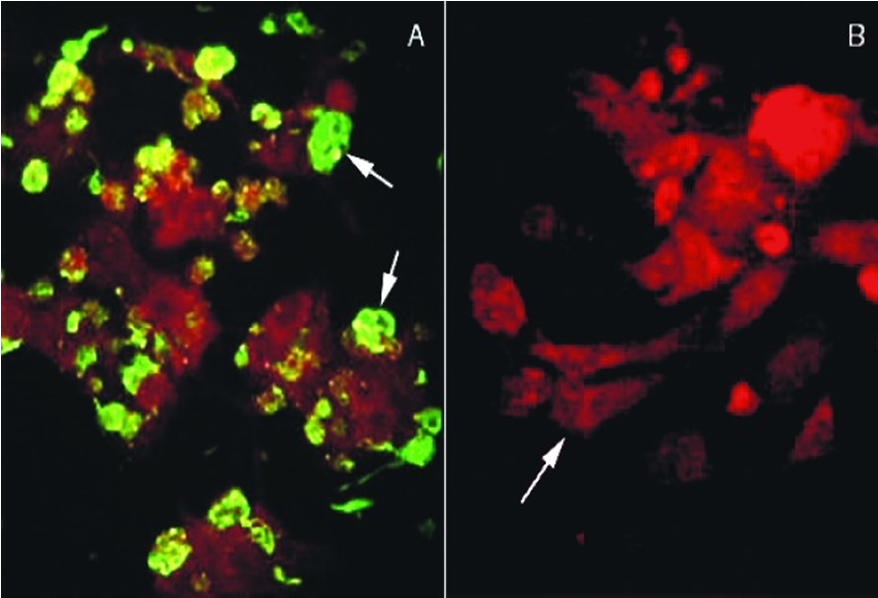
图 1显示的是感染犬瘟热病毒的足垫角质形成细胞的免疫荧光双染图片 [18] 。
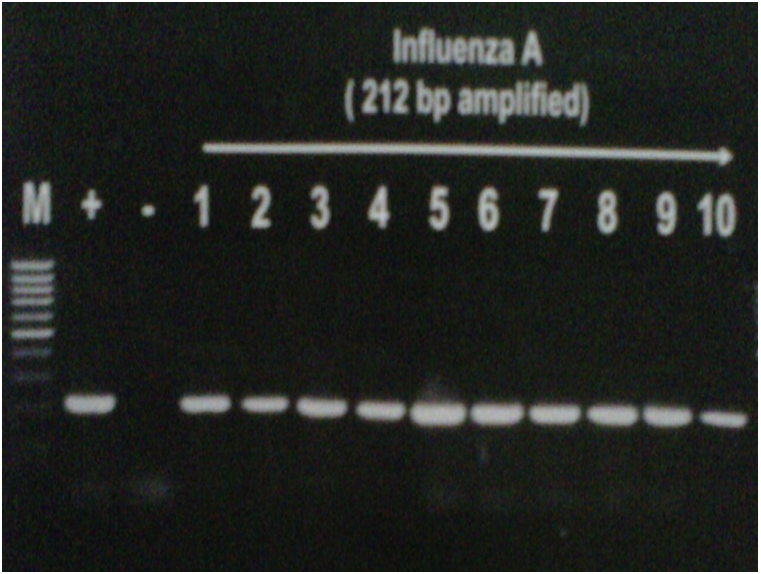
用分子技术从临床样本中直接对病毒基因组进行鉴定是21世纪的重大发现之一。核酸扩增技术包括PCR、基于核酸序列的扩增(NASBA)和劳伦斯利弗莫尔微生物检测阵列(LMDA)等都无疑是快速检测和鉴定大多数已知人类病毒的领先技术。PCR可以在 体外 将DNA序列的一段特定区域扩增 106倍,因此是一种极其敏感的检测手段。PCR还可以用于病毒RNA的鉴定,只需先将RNA逆转录成DNA,然后再进行PCR分析,这一方法被称之为逆转录PCR(RT-PCR)。自从Zhang和Evans 在1991年首次报道以来,陆续有人报道用RT-PCR方法进行流感病毒的鉴定 [19] 。图2显示的是用甲型流感病毒特异性的引物对流感病毒的分型进行鉴定(未发表数据)。 PCR的具体方法参见 Labome
用实时定量逆转录PCR(Real-time RT-PCR)来检测流感病毒比终点检测方法更为快捷,且敏感度跟细胞培养法相当甚至更佳 [20] 。图3用示意图显示了Real-time PCR的基本原理。之前有一项从儿童鼻分泌物中检测量呼吸道合胞病毒的研究表明,与免疫荧光和细胞培养法相比,Real-time PCR定量方法最为敏感 [21] 。然而,病毒的高突变率会使病毒核酸序列的发生剧烈变化,导致跟原来的PCR引物不匹配。因此,有需要发明一种快速且通用的病毒检测方法。而NASBA法虽然被认为有很高的敏感度,但由于自己制备NASBA反应混合液难度较高而商品化的试剂盒价格昂贵(见 Labome综述),该方法目前并没有被广泛应用。劳伦斯利弗莫尔国家实验室发明了一种新的用于微生物检测的分子生物学手段,它可以在同一个样本中对多种病毒类型进行快速检测。劳伦斯利弗莫尔微生物检测阵列(LLMDA)法用针对各种病毒基因组DNA的探针在24小时内即可完成病毒检测 [22, 23] 。另外,这些寡核苷酸探针还能够检测跟已知微生物具有同源性的新的物种 [23] 。关于LMDA法的检测过程参见 Labome 上的综述。
- 背景
病毒空斑试验是使用最广泛的病毒滴度检测方法之一 [24-28] 。Renato Dulbecco在1952年首先采用这种方法来计算噬菌体的感染力,自那以后,这种方法被广泛用于各种病毒的定量 [29-32] 。 一个蚀斑通常是由最初感染培养的宿主细胞单层的一个病毒颗粒形成的。 为此,在做空斑实验时,需要将病毒液作10倍梯度连续稀释,再分别感染相应的敏感细胞单层,孵育一段时间后,在细胞单层上覆盖一层半固体培养基(最常用的是琼脂糖)以防病毒从一开始的宿主细胞扩散至附近未感染的细胞。这样,每个病毒颗粒会在单层细胞上造成一个由未感染细胞围绕的小圆斑即称为空斑,当空斑长到足够大时,可在显微镜下观察甚至直接肉眼可见。 图4为病毒空斑试验的基本流程。计数不同稀释度下的空斑形成数量可以知道每毫升病毒颗粒数或每毫升空斑形成单位(PFU)。由于每个空斑来自于起初的1个病毒颗粒,这样可以从单个空斑中纯化得到来源于单个克隆的病毒种群。但很多时候,为了更清楚地辨别空斑,需要对细胞用MTT或中性红等染料进行染色以增加细胞与空斑间的对比度。另外,细胞状态对于空斑试验成功与否也至关重要,需要使用处于对数生长期且活力大于95%的健康的宿主细胞。病毒空斑试验比较费时,根据病毒种类不同,通常需要4-10天时间。还有,空斑试验只适用于那些能够繁殖并能感染培养细胞单层的病毒,以及可以破坏细胞的病毒。空斑试验的相对误差在10–100%,而 TCID50 的误差在35%左右 [33] 。
- 材料与设备
- 细胞生长培养基:含10%FBS(胎牛血清), 4-6mM Glutamine(谷氨酰胺)的高葡萄糖DMEM,若有需要可加入青霉素,链霉素,庆大霉素,两性霉素等
- 成斑培养基:含2%FBS(胎牛血清), 4-6 mM Glutamine(谷氨酰胺)的高葡萄糖DMEM
- 宿主细胞
- PBS磷酸缓冲液
- 琼脂糖,超纯
- 空斑染色材料(MTT或中性红)
- 细胞培养级无菌超纯水
- 移液枪头(10 到100 微升)
- 6孔细胞培养板
- 离心管
- 水浴箱
- 微波炉
- 倒置显微镜
- 生物安全柜(超净台)
- 二氧化碳培养箱
- 实验方案
- 细胞接种:每孔按1 x 106细胞每毫升培养基接种细胞至6-孔板里。轻轻地前后左右摇晃培养板使细胞分布均匀。将细胞置培养箱生长过夜。第二天,显微镜下观察细胞确认细胞是否分布均匀并达到80%以上的融合度。
- 制备琼脂糖:用水配制2%的琼脂糖,高压灭菌并使之溶化,然后将琼脂糖置于42°C水浴中使之保持在熔解状态。将细胞培养基预热至37°C。(1h后确认琼脂糖温度是否处于42°C,观察其是否仍处于溶解状态但又不至于烫手)。
- 准备病毒梯度稀释液:标记6个无菌离心管,用于制备病毒稀释液。在第1管内加入990μl,剩下5管分别加入900μl细胞生长培养基。按以下方法进行梯度稀释:在第1管中加入10μl病毒原液(稀释度1:100)充分混匀,然后从第1管吸100μl加入第2管,依次类推,进行10倍梯度稀释。管2至管6的稀释度分别为 10-3 到 10-7。
- 感染细胞单层:用无菌吸管吸去6孔板中的培养液,每孔加入100 μl上述稀释好的10-3 到 10-7 的病毒液,留一个孔不加作为空白对照。每板按同样的方法操作。室温放置1h让病毒进入宿主细胞。
- 覆盖琼脂糖:1h后,小心吸去病毒液,避免碰到细胞。将2%的琼脂糖和预热的培养基按1:1的比例混匀后加轻轻地加1.5mL至每孔细胞上,室温静置20min使其冷却凝固成覆盖层。将培养板置室温或 27°C 培养 6-10 日。
- 空斑观察和计数:用光从45度角照射培养板,或者可以将培养板倒置在一个黑色背景上,计数空斑数量。为了更易于观察,也可以每孔加入1mL 0.03%的中性红(用水或PBS稀释),室温或27°C 孵育2-3h,未被病毒感染的细胞会被中性红染上而中间未被着色的小区域即为空斑(直径约为0.5-3mm)。
计数每孔的空斑数量并按以下方法计算病毒滴度:病毒滴度(pfu/ml)=空斑数×(1 ml / 0.1 ml)/稀释倍数
- 背景
空斑试验对于确定病毒滴度非常有用,然而有几种类型的病毒在培养时不会形成空斑。这种情况下,它们的滴度可以用TCID50, LD50, EID50 等方法检测,这些病毒感染细胞后虽不至于造成细胞死亡,但会在一定时间(5-20天)后让细胞发生变性,称为致病变效应(CPE)。不过,并不是所有类型的病毒在组织培养时都会造成CPE的发生。因此,为了观察到致细胞病变效应,需注意要选择相匹配的病毒和细胞类型来进行该检测。半数组织感染量 TCID50 是使一半的单层细胞培养发生细胞病变的病毒量。获得的数据可以用几种统计学方法进行分析,例如Spearman-Karber分析或概率分析。在缺少实验材料或成本不够的情况下,TCID50检测法是很好的选择。虽然该检测法得到的病毒滴度的精确性和重复性不是很高,但可以很好地得到一个大致的滴度以及用于评价各处理组间的的差异 [27] 。空斑实验的相对误差有10-100%,而 TCID50 检测法为35%左右 [33] 。TCID50检测法在HIV-1、人类疱疹病毒、流感病毒等各种病毒的实验研究及临床诊断当中都有大量应用 [27, 34, 35] 。不过,TCID50 通常被认为比较耗时,因为它需要在培养的细胞中进行病毒感染,整个过程可能需要7天时间。以下是按Reed &Muench的方法检测TCID50 值的一个例子 [36] 。
- 材料与设备
- 细胞生长培养基:含10%FBS(胎牛血清), 4-6mM Glutamine(谷氨酰胺)的高葡萄糖DMEM,若有需要可加入青霉素,链霉素,庆大霉素,两性霉素等
- 维持培养基:上述新鲜的细胞培养基,但FBS浓度改为2%
- 宿主细胞
- 磷酸缓冲液(PBS)
- 移液枪头 (10 到100 微升)
- 48孔平底细胞培养板
- Falcon离心管
- 水浴箱
- 微波炉
- 倒置显微镜
- 生物安全柜(超净台)
- 二氧化碳培养箱
- 实验方案
- 宿主细胞接种:每孔按7x 104细胞每毫升培养基接种细胞至48-孔板里。轻轻地前后左右摇晃培养板使细胞分布均匀。将细胞置培养箱生长过夜。第二天,显微镜下观察细胞确认细胞是否分布均匀并达到80%以上的融合度。
- 制备病毒梯度稀释液:在病毒感染当天,将病毒原液用细胞生长培养基按以下方法做1:10梯度稀释。每个病毒样本标记24个无菌离心管,按6(行)×4(列)排好,每列有6管。在第一行的4个管内加入990μl培养基,剩下5行每管加900μl细胞生长培养基。将病毒储存液混匀后,加10μl至第1行的每个管中作1:100稀释。然后从第一行每管中吸取100μl加入第2行相对应的管中,依次类推,进行10倍梯度稀释。每列第2至第管6的稀释度分别为10-3 到 10-7.
- 感染细胞单层:在48-孔板的盖子上做好标记,每个条件有4个复孔,上方写上病毒名称,侧面标上每行相应的病毒稀释度,每板要有4孔不加病毒的阴性对照。小心吸去每孔内的培养基至剩0.1ml,轻轻加入0.1ml不同稀释度的病毒液至每孔内,每个稀释度感染4个复孔,按稀释度从高到低顺序加,即从48-孔板的下面往上加。在37°C孵育2h使病毒充分接触细胞(某些病毒如流感病毒在34°C生长更佳),然后在每孔中加入0.5ml维持培养基,并将细胞置37°C 或 34°C二氧化碳培养箱内培养1-4周,期间监测细胞病变CPE的形成情况。
- 观察并计算TCID50值:在倒置显微镜下观察各稀释度的CPE发生情况。按Reed &Muench提供的方法计算病毒滴度 [36] 。
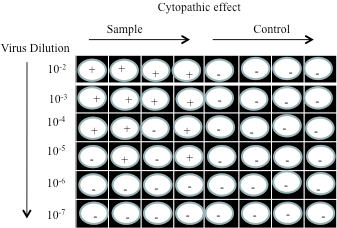
示例-图5为基于TCID50的终点稀释检测的一个示例。用一系列梯度稀释的病毒样本感染培养的细胞单层,每个稀释度感染4孔细胞(4个复孔)。培养一定的时间后,观察CPE,其中观察到CPE现象的孔标记为阳性(+)。根据图中显示,10-5 稀释度的4个孔中,有一半(2孔)的孔为CPE+,其余一半(2孔)为阴性。因此,该稀释度即为终点(50%的平行培养的细胞复孔被病毒感染的稀释度)。根据该结果可计算每毫升50%感染剂量(ID50 )。
相应的TCID50 滴度,可从耶鲁医学院的网上下载一个excel表格,并按照上面的方法进行计算 [37] 。
同样地,终点稀释法还可通过将病毒悬液感染动物来确定病毒的滴度。按照上面描述的的方法,类似地制备一系列梯度稀释的病毒液,然后将他们用于感染动物,而不是前面用的细胞。根据动物是否死亡或出现发烧、体重降低或瘫痪等临床症状来判断动物是否被病毒感染。如果最终后果是导致动物死亡或残废,相应地结果可用每毫升半数致死剂量(LD50)或每毫升半数致残剂量(PD50)来表示。
- 背景
IFA试验(又称为荧光灶试验FFA)是用培养的细胞株确定病毒滴度的一种快速检测方法,主要针对那些不能形成空斑或因为不导致明显的CPE效应而无法确定其TCID50的病毒。举个例子,甲肝病毒(HAV)在培养的细胞中不经多次传代的情况下,不产生明显的CPE [38] 。跟其它依赖病毒感染性的方法相比,IFA并不取决于病毒是否会导致细胞死亡,而是利用抗体染色的方法,最早在病毒感染细胞后2天即可对病毒进行检测。这在时间上要比其它感染试验比如标准的空斑实验及TCID50实验要缩短很多。IFA的结果用每毫升病灶形成单位(FFU/ml)来表示。IFA法在HIV-1、登革热病毒及流感病毒的实验研究及临床诊断当中有较多应用 [39-42] 。之前有项研究显示,跟实时定量PCR法相比,IFA法对于检测儿童鼻分泌物样本中的呼吸道合胞病毒(RSV)敏感度较低 [21] 。但是,另一项研究显示,跟空斑试验,TCID50及定量PCR相比, IFA对于检测水样中人类2型和41型腺病毒,JC多瘤病毒株Mad-4的敏感度和特异性都很高 [43] 。图6显示的是感染智利汉坦病毒CHI-7913的Vero E6细胞的IFA检测 [44] 。
总之,IFA被认为比传统的空斑形成试验和 TCID50试验快捷且敏感性更高,但由于需要用到抗体,该检测法的成本有时会偏高,而且有些抗体因为还没有商品化而很难获得,如果自己去制备抗体那代价会更高。另外,它还有一些缺点,比如不同的人计数得到的结果会有一定的差异,由于非特异结合导致染色的背景较高,或者与一些非病毒蛋白存在交叉反应等。图7A和7B分别是直接和间接IFA法的图示。
- 材料与设备
- 细胞生长培养基:含10%FBS(胎牛血清), 4-6mM Glutamine(谷氨酰胺)的高葡萄糖DMEM,若有需要可加入青霉素,链霉素,庆大霉素,两性霉素等
- 维持培养基:上述新鲜的细胞培养基,但FBS浓度改为2%
- 荧光标记一抗(直接IFA法)或无标记的一抗(间接IFA法)
- 荧光标记二抗(间接IFA法)
- 宿主细胞
- 磷酸缓冲液(PBS)
- 4%多聚甲醛(pH7.4)和TritonX-100或甲醇
- DAPI(染细胞核)或50%甘油(用水稀释)
- 移液枪头 (10 到100 微升)
- 48孔平底细胞培养板
- 离心管
- 荧光显微镜
- 超声仪
- 生物安全柜(超净台)
- 二氧化碳培养箱
- 实验方案
- 细胞接种:每孔按7x 104细胞每毫升培养基接种细胞至48-孔板里。轻轻地前后左右摇晃培养板使细胞分布均匀。将细胞置培养箱生长过夜。第二天,显微镜下观察细胞确认细胞是否分布均匀并达到80%以上的融合度。
- 制备病毒梯度稀释液:在病毒感染当天,将病毒原液用细胞生长培养基按以下方法做1:10梯度稀释。每个病毒样本标记24个无菌离心管,按6(行)×4(列)排好,每列有6管。在第一行的4个管内加入990μl培养基,剩下5行每管加900μl细胞生长培养基。将病毒储存液混匀后,加10μl至第1行的每个管中作1:100稀释。然后从第一行每管中吸取100μl加入第2行相对应的管中,依次类推,进行10倍梯度稀释。每列第2至第6管的稀释度分别为10-3 到 10-7。
- 感染细胞单层:在48-孔板的盖子上做好标记,每个条件有4个复孔,上方写上病毒名称,侧面标上每行相应的病毒稀释度,每板要有4孔不加病毒的阴性对照。小心吸去每孔内的培养基至剩0.1ml,轻轻加入0.1ml不同稀释度的病毒液至每孔内,每个稀释度感染4个复孔,按稀释度从高到低顺序加,即从48-孔板的下面往上加。在37°C 孵育2h使病毒充分接触细胞(某些病毒如流感病毒在37°C生长更佳),然后在每孔中加入0.5ml维持培养基,并将细胞置37°C 或 34°C 二氧化碳培养箱内培养。48-72 h后做免疫荧光染色。因为病毒要逐步扩散至相邻细胞以形成更多的感染灶,对于某些细胞这个过程可能需要4-5日。
- 染色,观察并计算FFU/ml:病毒感染48h后,吸去培养液,用预冷的甲醇固定细胞5min(或者用新鲜配制的4%PFA固定细胞,然后用0.2% triton X-100室温预渗透5min)。将细胞用PBS清洗后,如做直接IFA,跟推荐稀释度的荧光(Alexa fluor 488或 FITC)标记的一抗,若为间接IFA,跟未经标记的一抗孵育2h。吸去未结合的抗体,用PBS洗5遍,然后与荧光标记的二抗(间接IFA)进行孵育,再用PBS洗5遍,加入50%的甘油或DAPI,在荧光显微镜下观察结果。病毒滴度用每毫升荧光灶数量(IFU/ml)表示。
病毒滴度(IFU/ml) = 每孔荧光灶的平均数量X稀释度 X 体积指数(转换成ml)
PCR是用来检测病毒核苷酸的使用最广的方法之一。PCR分析也可以用于病毒RNA的鉴定,只需先将RNA逆转录成DNA,然后再进行PCR分析,这一方法被称之为逆转录PCR(RT-PCR)。实时PCR(或称为定量PCR,qPCR)是在反应过程中,实时对病毒核苷酸进行定量的方法。Real-time PCR有两种:一种是基于探针的叫TaqMan PCR,另一种是基于染料掺入的叫SYBR Green法。qPCR在HIV-1、登革热病毒及流感病毒的实验研究及临床诊断中有较多应用 [45-47] 。图3用示意图显示了Real-time PCR的基本原理。PCR技术的详细阐述请参见 Labome
免疫印迹和免疫沉淀法可以从受感染的细胞裂解液里检测各种病毒蛋白。
- 免疫印迹
免疫印迹(或叫Western blot)技术可以检测从细胞、组织、器官或体液里分离的特异的病毒蛋白。用去污剂提取细胞总蛋白进行SDS-PAGE电泳,将各种蛋白按分子量大小分开。然后通过转膜将蛋白转到硝酸纤维素膜上并跟抗体进行孵育,最后用一些极其敏感的指示分子(如碱性磷酸酶、辣根过氧化物酶等)进行显影观察。
自从发现人感染HIV-1逆转录病毒会引起艾滋病(AIDS)后,科学家们很快研制出了针对该病毒的敏感且特异的检测手段 [48] 。免疫印迹即为其中一种推荐的验证HIV感染的手段,特别是当酶免疫法无法得到一个明确的结果的时候。虽然免疫印迹的结果有些难以解释而且成本较高,使得该技术的应用有所减少,但该法仍然普遍地用于HIV-1的诊断、血清流行病学调查、血液捐献者筛选等。另外,免疫印迹还用于丙肝病毒抗体反应性的验证 ( CDC) [49, 50] 。近年来,该法还被认为是鉴定病毒颗粒蛋白组成的必要手段。通过对这些病毒蛋白进行分析可以更好地了解病毒感染机体的过程及其发病机理 [51, 52] 。有关免疫印迹技术的更多详细细节请参见 Labome。
- 免疫沉淀 (IP)
- 背景
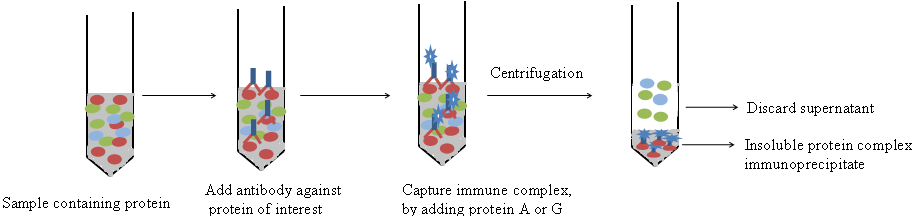
免疫沉淀技术(IP)主要是利用抗原与抗体之间的特异性结合,以及protein A/G与抗体IgG的Fc片段与之间的结合特性。做IP实验室,通常把protein A/G预先结合在琼脂糖珠上,使之与抗体及含目的蛋白的样本溶液进行反应,交联在琼脂糖珠上的protein A/G就可以吸附目的蛋白,通过低速离心即可将目的蛋白从蛋白混合溶液分离出来。分离得到的样本随即可以通过SDS-PAGE分析来确定蛋白的分子量及含量,研究蛋白与蛋白相互作用,以及了解蛋白翻译后修饰情况。由于用免疫沉淀分离得到的样本常常混有很多其它背景蛋白,直接用蛋白组学分析来鉴定免疫沉淀得到的蛋白并不太可行,但对于那些特异性很高的抗体,尤其是在这些特异的抗体被预先共价交联至固体载体小珠上不会被洗脱的情况下,还是可以用蛋白组学方法来鉴定分离得到的蛋白,因为这样可以减少最后洗脱下来的样本中抗体的含量 [53] 。举个免疫沉淀法应用的例子,该法被广泛用于I型单纯疱疹病毒(HSV-1)蛋白ICP8 和 ICP27相互作用的鉴定 [54, 55] 。类似的,还有研究创新性的用protein A琼脂糖珠从细胞碎片中共沉淀得到BTV-10 [56] 。这些基于免疫沉淀的研究还林林总总的被用于各类病毒包括呼吸道合胞病毒(RSV)、埃博拉病毒、流感病毒及丙型肝炎病毒(HCV)的实验研究及诊断 [57-60] 。另外,把免疫沉淀和免疫印迹技术相结合,可以很好地克服单单采用其中一项技术存在的一些缺点 [61] 。例如,先用免疫沉淀富集感兴趣的蛋白,可以大大地提高免疫印迹检测目的蛋白的特异性和敏感度 [61] 。最近,免疫沉淀-印迹方法被成功的用于一个新的自身抗体anti-p155的特性鉴定,以及用于病人血清样本中该自身抗体的检测以研究其临床联系 [61] 。
- 材料与设备
- 一抗
- Protein A 或Protein G琼脂糖珠:按照厂家说明书操作。兔IgG用Protein A,小鼠IgG用Protein G
- 细胞裂解液:用之前新鲜加入1 mM PMSF
- 3X SDS上样缓冲液:Tris-HCl (pH 6.8, 25°C), 6% SDS(w/v,重量体积比), 30% 甘油, 二硫苏糖醇(DTT), 溴酚蓝
- 磷酸缓冲液
- 10 cm2 细胞培养皿
- 离心管
- 移液枪头 (10 到100 微升)
- 细胞刮檫
- 超声仪
- 离心机
- 实验方案
- 1. 裂解细胞:吸去细胞培养液,加预冷PBS洗1遍,吸去PBS,每个10 cm2细胞培养皿加入0.5mL预冷的1X细胞裂解液,置冰上裂解5min,用细胞刮擦将细胞从培养板上刮下并将裂解液收集到离心管内,整个过程在冰上操作。将样本在冰浴中超声3次,然后高速离心(14,000 X g,10min),收集上清。
- 2. 免疫沉淀:取200 μl细胞裂解液,加入一定稀释度的一抗(起始范围可用1-10 ug),于4°C旋转混匀孵育过夜后加入50μl Protein A 或Protein G琼脂糖珠,4°C旋转混匀孵育1-3h,在4°C ,3000 X rpm离心30s,将protein A/G琼脂糖珠离心至管底,将上清小心吸去,protein A/G琼脂糖珠沉淀用500 μl 1 X细胞裂解液洗2-4次,整个过程在冰上操作,最后将protein A/G琼脂糖珠用20μl的3×SDS 上样缓冲液重悬,95–100°C 煮5-10min,随后进行SDS-PAGE和免疫印迹分析。详细方法参见 Labome。
- 背景
- 背景
酶联免疫吸附试验(ELISAs) (也叫EIA) 的基础是通过抗原抗体的酶标记,该法很好地融合了酶反应的敏感度和抗体的特异性。用ELISA法从细胞、组织、器官或体液中检测特定的病毒蛋白比免疫印迹法要更加快速。ELISA法主要有两大类型:检测抗原的ELISA(检测病毒蛋白),是将特异性抗体与固相载体结合去检测样品中相应的病毒抗原;而抗体检测ELISA是将抗原包被于固相基质表面用以检测样本中的抗体水平。图10显示了该两种ELISA方法的工作原理。通常来讲,ELISA技术敏感度很高,可以用于检测含量很低,皮摩尔到纳摩尔水平(10-12 到 10-9 mol/L)的蛋白。 ELISA是目前检测HIV-1病毒抗体(见 疾控中心资料)中使用最广的血清学实验之一。ELISA是检测流感病毒抗原非常有用的诊断方法,它比其他传统的病毒检测方法要快速得多 [62] 。还有一项研究也证明:ELISA对于甲型流感病毒的快速检测与鉴定,跟传统的方法相比具有明显的优越性 [63] 。研究者发明了一种简易但标准化的中和抗体酶免疫分析 (Nt-EIA) 方法来检测感染麻疹病毒的Vero细胞中麻疹病毒中和抗体的含量 [64] 。丙肝病毒诊断的酶免疫分析新方法也在不断的被验证当中 [65, 66] 。ELISA法正被广泛用于HIV-1、登革热、流感等病毒的实验研究与诊断 [67-69] 。虽然ELISA比传统的病毒空斑试验或 TCID50测定方法要快速,但它的试剂价格昂贵使得检测成本较高,有时还会缺乏商品化的抗体,而自己制备抗体成本过高。另外,由于非特异结合或与非病毒来源的蛋白交叉反应导致的检测背景过高也会影响结果的一致性。
- 材料与设备
- 包被液(50 mM碳酸钠,pH 9.6; 20 mMTris-HCL, pH 8.5)
- 洗涤液(PBS-Tween):没升PBS加1 ml Tween-20
- 封闭液 (含1% BSA 的PBS 或脱脂牛奶)
- 一抗和二抗(用封闭液稀释)
- 溶于0.1M醋酸钠的TMB溶液 (加30%的双氧水使醋酸的终浓度为0.01%)
- 移液枪头 (10 到100 微升)
- 高吸附平底96孔培养板((Nunc™)
- 离心管
- 酶标仪
- 实验方案: 抗原检测 ELISA(双抗夹心法)
- 抗体包被:用包被液稀释抗体(浓度范围为0.2-10 µg/ml),加100µl/孔至Nunc™高吸附平底96孔培养板,用封口膜将培养板密封以防止挥发,置于4°C冰箱孵育过夜或置室温孵育2 h(也可以在37°C 孵育 1 h)。第二天,弃去包被液并用蒸馏水或去离子水洗两遍。
- 封闭:每孔加入50-200µl封闭液,37°C或室温孵育1 h,孵育时用保鲜膜包好或置于湿盒中。
- 加受检样本(病毒抗原):弃去封闭液,将含病毒的样本用封闭液稀释至合适浓度后加100µl 至每孔,室温或4°C孵育2-4 h。吸去含检测样本的溶液,用洗涤液或蒸馏水清洗至少3遍。
- 加检测抗体:按照说明书推荐方法稀释酶标抗体至合适浓度,每孔加入200µl,室温孵育30 min后,吸弃抗体,用洗涤液洗3遍,加入一定量的TBM底物溶液反应30 min后加入终止液终止反应。在酶标仪上检测相应波长的吸光度。
- 实验方案: 抗体检测 ELISA
- 抗原包被:用包被液稀释抗原(浓度范围为0.2-10 µg/ml),加100µl/孔至Nunc™高吸附平底96孔培养板,用封口膜将培养板密封以防止挥发,置于4°C冰箱孵育过夜或置室温孵育2 h(也可以在37°C 孵育 1 h)。第二天,弃去包被液并用蒸馏水或去离子水洗两遍。
- 封闭:每孔加入50-200µl封闭液,37°C或室温孵育1 h,孵育时用保鲜膜包好或置于湿盒中。
- 加受检抗体:弃去封闭液,加100µl含抗病毒抗体的样本至每孔,37°C孵育1 h。抗体样本的推荐稀释度为1:10, 1:100, 1:1000, 1:10,000 以及不稀释。吸去含被检抗体样本的溶液,用洗涤液或蒸馏水清洗10遍。用封闭液将二抗稀释至厂家推荐浓度,每孔加入100 µl,密封,在室温或37°C摇晃孵育1 h,洗涤。
- 底物显色:每孔加100 µl TBM底物溶液,室温反应5, 15或30 min(通常为30min)。如有需要,当颜色深度达到要求后,加入50µl 10%的磷酸终止反应,在酶标仪上检测相应波长的吸光度。
- 背景
血凝试验是对病毒颗粒进行间接定量的最常用的方法,检测样本通常为细胞培养上清或从鸡蛋中收集的鸡胚尿囊液。这个实验是利用有些病毒蛋白(如流感病毒的凝集素)具有结合并凝集红细胞(RBC)的特性。自40年代时Salk研究所的科学家对该方法做了几个关键的改进后,这种利用凝集特性来检测病毒颗粒数量的血凝试验的方法一直沿用至今,基本上没什么大的改动 [70-72] 。尽管原理简单,该方法样本制备过程比较费力,存在一些缺点。例如,由于红细胞凝集能力会随着时间增长而下降,为了保证结果的重复性,红细胞每次必需新鲜制备。另外,每次红细胞的来源不同,因此还需要用参考标准对实验进行标准化。还有,由于该试验不需要病毒复制过程,不像其它病毒定量方法(如空斑实验或 TCID50法),血凝试验不能对病毒的感染能力进行评估。在检测时,悬液中的红细胞会沉积至培养孔的底部,呈点状分布,但如果检测样本中的病毒表面带有凝集素的话,可以使红细胞全凝集在底部,不会被重悬。通过加入一定量的红细胞和不同稀释度的病毒液,可以对病毒滴度进行估算。图11为标准凝集试验的原理图。
- 材料和仪器
- 火鸡红细胞
- 磷酸缓冲液(PBS)
- 15 mL离心管
- 移液枪头(10-100微升)
- 一次性移液管(1 ml, 5 ml, 10 ml)
- 圆底96孔板
- 离心机
- 生物安全柜
- 实验方案
- 制备病毒稀释液加50μl/孔PBS至圆底96孔板,加50μl病毒液至第一孔,混合均匀后,吸50L至第2孔混匀,依次倍比稀释,最后一孔吸50L,弃至漂白水溶液中。依次向各孔加入0.5% 火鸡红细胞悬液50L,轻轻混匀,室温下静置30-60min后观察结果。阴性结果即不凝集者(-),红细胞呈点状沉于孔底,若红细胞发生凝集,红细胞会均匀平铺于整个孔底,则为阳性。
- 结果计算:计数格子形成数量然后按照Donald 和 Isaacs描述的方法计算病毒滴度 [73] 。病毒浓度(病毒粒子/ml)= 107 x HA滴度。
- 背景
通过流式细胞仪分析来进行病毒定量最先应用于检测昆虫细胞中的杆状病毒 [74] ,而后,Brussaard等成功地用流式细胞仪分析各类形态和基因组大小各异的病毒 [75] 。研究显示,流式细胞仪法检测病毒的结果与荧光显微镜检测结果相当。SYBR Green I 或SYBR Gold被广泛用于病毒的流式细胞仪计数 [76, 77] 。SYBR Green I是一种非常敏感的能结合双链DNA(dsDNA)和RNA的绿色荧光染料。虽然DAPI也被用于流式病毒定量,但这个染料的病毒颗粒染色比较暗淡,不易计数。相反,SYBR Green I的荧光非常亮且背景低,是一个优选选择。FCM被成功用于实验室生物样本的病毒计数 [78, 79] 。最近,Gates等发展了一种新的FCM法用于检测MRC-5细胞水痘 - 带状疱疹病毒(VZV)的减毒疫苗株(vOka/Merck)的感染性,该法比空斑试验更高效 [80] 。另外还研制出了一些用于检测HIV-1病毒的FCM检测方法 [81, 82] 。FCM法也被用于定量昆虫细胞中产生的杆状病毒颗粒 [83] 。Sivaraman D等建议FCM可用于多种病毒感染的实际诊断 [84] 。跟荧光显微镜法相比,FCM法可在单细胞水平获得统计学上可靠的数据,且花费的精力相对较少。FCM得到的荧光分布结果还可进一步用于病毒蛋白合成及病毒颗粒释放等数据的统计估算和数学建模 [85] 。最近发展了一种称为荧光共振能量转移(FRET)的方法,即在细胞中表达一对相互间可激发能量转移的荧光蛋白,用于快速检测病毒的感染力。FCM可以检测荧光蛋白间的重叠信号,因此可以用于检测FRET信号。FCM可以快速地检测每个病毒感染的细胞上的荧光信号数量 [86-88] 。一般来讲,FCM病毒颗粒检测法敏感度不高 [78, 79] ,但某些商品化的FCM方法(如Virus Counter® 2100)可以简单快速地进行病毒定量且敏感度较高。在病毒计数时,每个样本先用两种不同的荧光染料染色:一个特异的染核酸,另一个染蛋白,孵育30min后,将染色样本置于病毒计数仪器上分析以计算病毒颗粒的浓度(vp/mL)。
- 材料和仪器
- 电镜级别的戊二醛
- SYBR® green I染色工作液(母液为10,000×,溶于DMSO)
- 无菌TE溶液,pH8.0
- 超纯水(Milli-Q水)
- 含0.5% BSA的PBS(PBSA)
- 液氮
- 离心管
- 超声仪
- 流式细胞仪,PASIII (Partec公司), CyFlow® Space (Partec公司), Cytomics FC 500, MPL (Beckman Coulter公司)
- 实验方案
- 制备系列病毒稀释液:标记6个无菌离心管,每管加入270 μl PBSA,加30μl病毒母液至第一管,混匀,然后取30μl至第2管,依次类推,制备10倍梯度稀释液。FCM分析需要跟病毒浓度的对数建立的线性关系,其检测下限为105病毒颗粒每毫升 [83] 。
- 病毒样本固定,染色和分析:用0.5%戊二醛在4°C避光固定样本15-30 min,将样本用-180 °C液氮冷冻处理1 min后重新溶解以预渗透样本,最后用Tris-EDTA (TE)稀释。加入5 μL 100x溶于DMSO的SYBR® Green I,在80°C避光染色10 min,超声30 min,进行FCM分析。绿色荧光光电倍增管(PTM)调至最大电压,以去除电或激光噪音干扰。检测病毒样本前还需要使用空白对照,由TE溶液和经0.2-μm滤器或30-kDa超滤管过滤的样本按病毒溶液同样稀释度配制。空白对照样本的稀释、染色和处理应该跟病毒样本一致。只有当空白对照的阳性数量((0–15 events s–1)及荧光背景很低时才能进行样本检测。FCM法的详细流程请参见 Labome。
- 背景
大部分病毒颗粒都非常小无法在光镜下直接观察,只能通过透射电镜(TEM)进行观察。在1948年,科学家首次用透射电镜对天花和水痘病毒进行区分 [89], 此后一段时间电镜被广泛用于病毒的分类,尤其是应用电镜负染技术发现了很多肠道病毒 [90, 91] 。虽然透射电镜法目前正逐渐被敏感度更高的PCR和免疫荧光等方法所取代,但在病毒学研究的某些方面,如新病毒的发现,病毒特征描述及滴度测定等,透射电镜仍然十分必要。使用透射电镜的一个主要优点是它不需要各种病毒特异性的检测试剂;这在致病原不明的病毒大爆发时尤为重要,因为如果致病病毒种类未知,就无法选择针对性的检测试剂和检测手段。Goldsmith 和 Miller撰写了一篇综述细致地描述了电镜法检测病毒的新方法 [92] 。负染透射电镜技术一直是埃博拉病毒、立百病毒(Hendra and Nipah)、SARS病毒等新病毒发现和鉴定的重要工具 [93-97]. 一次猴痘病毒在人群中造成爆发时,美国采用透射电镜首次鉴定出了该病毒 [98] 。另外一篇Roingeard在2008年的综述描述了透射电镜检测病毒的现状及前景展望 [99] 。
如果预计样本中病毒数量较多(>107 病毒颗粒/ml),可以直接用负染法透射电镜进行观察,但如果预计样本中病毒的滴度较低,病毒样本可以先用蔗糖密度梯度或超离进行浓缩纯化。这两种病毒浓缩方法虽然可以提高检测范围,将病毒的检测下限降至~105病毒颗粒/ml,但它也存在几个局限性,如蔗糖可以破坏一些极不稳定的病毒颗粒,而超离可能将碎片与病毒颗粒一起浓缩导致检测限度下降。负染的方法有好几种,用的较多的是使用0.05-2%乙酸铀酰(uranyl acetate),1-2%磷钨酸(phosphotungstic acid,PTA)和0.05-5%钼酸铵(ammonium molybdate) [92] 。负染透射电镜的图片可以直接显示单个病毒颗粒的形态并可以通过计数病毒颗粒数量确定病毒的浓度。然而,由于仪器使用成本高、需要的空间及设施较多,透射电镜分析只在某些有条件的中心才能开展。
- 材料和仪器
- 辉光放电装置
- 电镜网格镊子(2把以上)
- 乙酸铀酰(Uranyl acetate)
- 超纯水 (如. Milli-Q 水)
- EM网格
- Petri 培养皿
- 废液回收罐(回收乙酸铀酰)
- 透射电镜
- 实验方案
将病毒吸附至透射电镜网格:用镊子夹住网格,滴5 μl病毒液至网格的光面让其吸附3 min左右后,马上将网格置于2%乙酸铀酰中染色3遍,每次持续45s。
图 12显示的是负染的裸露的二十面体腺病毒颗粒的透射电镜图片 [92] 。
很多图谱、其他的网站还有博客里都能找到很好的病毒负染电镜的图片,例如virology.net 以及参考文献 [100] 。
过去几十年里,为了定量表征病毒样本发明了很多检测方法。传统的用于定量具有感染力的病毒颗粒数量的方法比较费时费力,且往往重复性不高。免疫荧光法比空斑试验和TCID 50实验快速,但由于需要使用抗原特异性的抗体使得成本较高。另外一方面,那些检测病毒抗原或病毒基因表达的方法通常比较快速且重复性好,但不能对病毒颗粒进行精确计数。同样的,qPCR可以在几个小时内得到结果,但也比较费力,需要技术熟练的操作者,而且极易导致污染。透射电镜定量法虽然对于确定病毒的形状和病毒颗粒总数精确度较高,但比较费时,成本很高且在样本数量多时可行性不高,加上透射电镜样本制备过程也很费事,该技术需要精密的仪器和熟练的操作者。为了克服目前检测方法中的这些局限性,仍然有必要发展新的低成本高效益的分析方法,让使用者们可以简单快速的确定病毒的浓度,然而任何新的技术都需要对其效率、精度和线性范围进行仔细的评估。
- Enders J. Cytopathology of virus infections: particular reference to tissue culture studies. Annu Rev Microbiol. 1954;8:473-502 pubmed
- Leland D, Ginocchio C. Role of cell culture for virus detection in the age of technology. Clin Microbiol Rev. 2007;20:49-78 pubmed
- Hsiung G. Diagnostic virology: from animals to automation. Yale J Biol Med. 1984;57:727-33 pubmed
- Forman M S, Valsamakis A. Specimen collection, transport, and processing: virology. In: Murray PR, Baron EJ, Jorgensen JH, Pfaller MA, Yolken RH editors. Manual of clinical microbiology. 8th ed. ASM Press: Washington, DC; 2003. AND Smith TF. Specimen requirements: selection, collection, transport, and processing. In: Specter S, Hodinka RL, Young SA editors. Clinical virology manual, 3rd ed. ASM Press: Washington, DC; 2000.
- Gleaves C, Smith T, Shuster E, Pearson G. Rapid detection of cytomegalovirus in MRC-5 cells inoculated with urine specimens by using low-speed centrifugation and monoclonal antibody to an early antigen. J Clin Microbiol. 1984;19:917-9 pubmed
- Rodriguez Roche R, Alvarez M, Guzman M, Morier L, Kouri G. Comparison of rapid centrifugation assay with conventional tissue culture method for isolation of dengue 2 virus in C6/36-HT cells. J Clin Microbiol. 2000;38:3508-10 pubmed
- Espy M, Smith T, Harmon M, Kendal A. Rapid detection of influenza virus by shell vial assay with monoclonal antibodies. J Clin Microbiol. 1986;24:677-9 pubmed
- Navarro-Marí J, Sanbonmatsu-Gámez S, Pérez-Ruiz M, De La Rosa-Fraile M. Rapid detection of respiratory viruses by shell vial assay using simultaneous culture of HEp-2, LLC-MK2, and MDCK cells in a single vial. J Clin Microbiol. 1999;37:2346-7 pubmed
- Anestad G. Surveillance of respiratory viral infections by rapid immunofluorescence diagnosis, with emphasis on virus interference. Epidemiol Infect. 1987;99:523-31 pubmed
- Daisy J, Lief F, Friedman H. Rapid diagnosis of influenza A infection by direct immunofluorescence of nasopharyngeal aspirates in adults. J Clin Microbiol. 1979;9:688-92 pubmed
- Fauvel M, Ozanne G. Immunofluorescence assay for human immunodeficiency virus antibody: investigation of cell fixation for virus inactivation and antigen preservation. J Clin Microbiol. 1989;27:1810-3 pubmed
- Klespies S, Cebula D, Kelley C, Galehouse D, Maurer C. Detection of enteroviruses from clinical specimens by spin amplification shell vial culture and monoclonal antibody assay. J Clin Microbiol. 1996;34:1465-7 pubmed
- Coffin S, Hodinka R. Utility of direct immunofluorescence and virus culture for detection of varicella-zoster virus in skin lesions. J Clin Microbiol. 1995;33:2792-5 pubmed
- Pouletty P, Chomel J, Thouvenot D, Catalan F, Rabillon V, Kadouche J. Detection of herpes simplex virus in direct specimens by immunofluorescence assay using a monoclonal antibody. J Clin Microbiol. 1987;25:958-9 pubmed
- Liu I, Chen P, Yeh S, Chiang Y, Huang L, Chang M, et al. Immunofluorescence assay for detection of the nucleocapsid antigen of the severe acute respiratory syndrome (SARS)-associated coronavirus in cells derived from throat wash samples of patients with SARS. J Clin Microbiol. 2005;43:2444-8 pubmed
- Engelhardt P, Wyder M, Zurbriggen A, Grone A. Canine distemper virus associated proliferation of canine footpad keratinocytes in vitro. Vet Microbiol. 2005;107:1-12 pubmed
- Zhang W, Evans D. Detection and identification of human influenza viruses by the polymerase chain reaction. J Virol Methods. 1991;33:165-89 pubmed
- van Elden L, Nijhuis M, Schipper P, Schuurman R, van Loon A. Simultaneous detection of influenza viruses A and B using real-time quantitative PCR. J Clin Microbiol. 2001;39:196-200 pubmed
- Gueudin M, Vabret A, Petitjean J, Gouarin S, Brouard J, Freymuth F. Quantitation of respiratory syncytial virus RNA in nasal aspirates of children by real-time RT-PCR assay. J Virol Methods. 2003;109:39-45 pubmed
- Cooper L, Scott T. Differential evolution of eastern equine encephalitis virus populations in response to host cell type. Genetics. 2001;157:1403-12 pubmed
- López-Bueno A, Mateu M, Almendral J. High mutant frequency in populations of a DNA virus allows evasion from antibody therapy in an immunodeficient host. J Virol. 2003;77:2701-8 pubmed
- LaBarre D, Lowy R. Improvements in methods for calculating virus titer estimates from TCID50 and plaque assays. J Virol Methods. 2001;96:107-26 pubmed
- Takenaka A, Gibbs C, Gajdusek D. Antiviral neutralizing antibody to Hantaan virus as determined by plaque reduction technique. Arch Virol. 1985;84:197-206 pubmed
- Dulbecco R. Production of Plaques in Monolayer Tissue Cultures by Single Particles of an Animal Virus. Proc Natl Acad Sci U S A. 1952;38:747-52 pubmed
- Biacchesi S, Skiadopoulos M, Yang L, Murphy B, Collins P, Buchholz U. Rapid human metapneumovirus microneutralization assay based on green fluorescent protein expression. J Virol Methods. 2005;128:192-7 pubmed
- Yale School of Medicine Infectivity Calculator. 来自: www.med.yale.edu/micropath/pdf/Infectivity%20calculator.xls
- Cromeans T, Sobsey M, Fields H. Development of a plaque assay for a cytopathic, rapidly replicating isolate of hepatitis A virus. J Med Virol. 1987;22:45-56 pubmed
- Payne A, Binduga-Gajewska I, Kauffman E, Kramer L. Quantitation of flaviviruses by fluorescent focus assay. J Virol Methods. 2006;134:183-9 pubmed
- Schoepp R, Beaty B. Titration of dengue viruses by immunofluorescence in microtiter plates. J Clin Microbiol. 1984;20:1017-9 pubmed
- Kvinesdal B, Nielsen C, Poulsen A, Højlyng N. Immunofluorescence assay for detection of antibodies to human immunodeficiency virus type 2. J Clin Microbiol. 1989;27:2502-4 pubmed
- Galeno H, Mora J, Villagra E, Fernandez J, Hernandez J, Mertz G, et al. First human isolate of Hantavirus (Andes virus) in the Americas. Emerg Infect Dis. 2002;8:657-61 pubmed
- Schochetman G, Epstein J, Zuck T. Serodiagnosis of infection with the AIDS virus and other human retroviruses. Annu Rev Microbiol. 1989;43:629-59 pubmed
- Damen M, Zaaijer H, Cuypers H, Vrielink H, van der Poel C, Reesink H, et al. Reliability of the third-generation recombinant immunoblot assay for hepatitis C virus. Transfusion. 1995;35:745-9 pubmed
- http. 来自: www.hepcnet.net/serologic_tests.html
- Kong Q, Xue C, Ren X, Zhang C, Li L, Shu D, et al. Proteomic analysis of purified coronavirus infectious bronchitis virus particles. Proteome Sci. 2010;8:29 pubmed
- Ren X, Xue C, Kong Q, Zhang C, Bi Y, Cao Y. Proteomic analysis of purified Newcastle disease virus particles. Proteome Sci. 2012;10:32 pubmed
- Maxwell K, Frappier L. Viral proteomics. Microbiol Mol Biol Rev. 2007;71:398-411 pubmed
- Fontaine-Rodriguez E, Taylor T, Olesky M, Knipe D. Proteomics of herpes simplex virus infected cell protein 27: association with translation initiation factors. Virology. 2004;330:487-92 pubmed
- Taylor T, Knipe D. Proteomics of herpes simplex virus replication compartments: association of cellular DNA replication, repair, recombination, and chromatin remodeling proteins with ICP8. J Virol. 2004;78:5856-66 pubmed
- Bernstein J, Hruska J. Respiratory syncytial virus proteins: identification by immunoprecipitation. J Virol. 1981;38:278-85 pubmed
- Johnson R, McCarthy S, Godlewski P, Harty R. Ebola virus VP35-VP40 interaction is sufficient for packaging 3E-5E minigenome RNA into virus-like particles. J Virol. 2006;80:5135-44 pubmed
- Waxman L, Whitney M, Pollok B, Kuo L, Darke P. Host cell factor requirement for hepatitis C virus enzyme maturation. Proc Natl Acad Sci U S A. 2001;98:13931-5 pubmed
- Khanna M, Kumar P, Chugh L, Prasad A, Chhabra S. Evaluation of influenza virus detection by direct enzyme immunoassay (EIA) and conventional methods in asthmatic patients. J Commun Dis. 2001;33:163-9 pubmed
- Waner J, Todd S, Shalaby H, Murphy P, Wall L. Comparison of Directigen FLU-A with viral isolation and direct immunofluorescence for the rapid detection and identification of influenza A virus. J Clin Microbiol. 1991;29:479-82 pubmed
- Lee M, Cohen B, Hand J, Nokes D. A simplified and standardized neutralization enzyme immunoassay for the quantification of measles neutralizing antibody. J Virol Methods. 1999;78:209-17 pubmed
- Niu X, Wang X, Liu G, Li Y. [Establishment of the evaluation reference system for domestic anti-hepatitis C virus diagnostic enzyme immunoassay kits]. Xi Bao Yu Fen Zi Mian Yi Xue Za Zhi. 2013;29:761-4 pubmed
- Filice G, Soldini L, Orsolini P, Razzini E, Gulminetti R, Campisi D, et al. Sensitivity and specificity of anti-HIV ELISA employing recombinant (p24, p66, gp120) and synthetic (gp41) viral antigenic peptides. Microbiologica. 1991;14:185-94 pubmed
- de Boer G, Back W, Osterhaus A. An ELISA for detection of antibodies against influenza A nucleoprotein in humans and various animal species. Arch Virol. 1990;115:47-61 pubmed
- Cuzzubbo A, Vaughn D, Nisalak A, Solomon T, Kalayanarooj S, Aaskov J, et al. Comparison of PanBio dengue duo enzyme-linked immunosorbent assay (ELISA) and MRL dengue fever virus immunoglobulin M capture ELISA for diagnosis of dengue virus infections in Southeast Asia. Clin Diagn Lab Immunol. 1999;6:705-12 pubmed
- Hirst G. THE QUANTITATIVE DETERMINATION OF INFLUENZA VIRUS AND ANTIBODIES BY MEANS OF RED CELL AGGLUTINATION. J Exp Med. 1942;75:49-64 pubmed
- DONALD H, Isaacs A. Counts of influenza virus particles. J Gen Microbiol. 1954;10:457-64 pubmed
- Stoffel C, Kathy R, Rowlen K. Design and characterization of a compact dual channel virus counter. Cytometry A. 2005;65:140-7 pubmed
- Brussaard C, Marie D, Bratbak G. Flow cytometric detection of viruses. J Virol Methods. 2000;85:175-82 pubmed
- Marie -, Brussaard -, Thyrhaug -, Bratbak -, Vaulot -. Enumeration of marine viruses in culture and natural samples by flow cytometry. Appl Environ Microbiol. 1999;65:45-52 pubmed
- Chen F, Lu J, Binder B, Liu Y, Hodson R. Application of digital image analysis and flow cytometry to enumerate marine viruses stained with SYBR gold. Appl Environ Microbiol. 2001;67:539-45 pubmed
- Yan X, Zhong W, Tang A, Schielke E, Hang W, Nolan J. Multiplexed flow cytometric immunoassay for influenza virus detection and differentiation. Anal Chem. 2005;77:7673-8 pubmed
- Hwang Y, Chen W, Yates M. Use of fluorescence resonance energy transfer for rapid detection of enteroviral infection in vivo. Appl Environ Microbiol. 2006;72:3710-5 pubmed
- Wunschmann S, Stapleton J. Fluorescence-based quantitative methods for detecting human immunodeficiency virus type 1-induced syncytia. J Clin Microbiol. 2000;38:3055-60 pubmed
- Shen C, Meghrous J, Kamen A. Quantitation of baculovirus particles by flow cytometry. J Virol Methods. 2002;105:321-30 pubmed
- Lambeth C, White L, Johnston R, de Silva A. Flow cytometry-based assay for titrating dengue virus. J Clin Microbiol. 2005;43:3267-72 pubmed
- Kao C, Wu M, Chiu Y, Lin J, Wu Y, Yueh Y, et al. Flow cytometry compared with indirect immunofluorescence for rapid detection of dengue virus type 1 after amplification in tissue culture. J Clin Microbiol. 2001;39:3672-7 pubmed
- Sydow F, Santiago M, Neves-Souza P, Cerqueira D, Gouvea A, Lavatori M, et al. Comparison of dengue infection in human mononuclear leukocytes with mosquito C6/36 and mammalian Vero cells using flow cytometry to detect virus antigen. Mem Inst Oswaldo Cruz. 2000;95:483-9 pubmed
- Nagler F, Rake G. The Use of the Electron Microscope in Diagnosis of Variola, Vaccinia, and Varicella. J Bacteriol. 1948;55:45-51 pubmed
- Gust I, Kaldor J, Cross G, Waugh M, Ferris A. Virus-like particles associated with a faecal antigen from hepatitis patients and with Australia antigen. Aust J Exp Biol Med Sci. 1971;49:1-9 pubmed
- Kapikian A, Wyatt R, Dolin R, Thornhill T, Kalica A, Chanock R. Visualization by immune electron microscopy of a 27-nm particle associated with acute infectious nonbacterial gastroenteritis. J Virol. 1972;10:1075-81 pubmed
- Johnson K, Lange J, Webb P, Murphy F. Isolation and partial characterisation of a new virus causing acute haemorrhagic fever in Zaire. Lancet. 1977;1:569-71 pubmed
- Pattyn S, van der Groen G, Courteille G, Jacob W, Piot P. Isolation of Marburg-like virus from a case of haemorrhagic fever in Zaire. Lancet. 1977;1:573-4 pubmed
- Chua K, Wong E, Cropp B, Hyatt A. Role of electron microscopy in Nipah virus outbreak investigation and control. Med J Malaysia. 2007;62:139-42 pubmed
- Hyatt A, Zaki S, Goldsmith C, Wise T, Hengstberger S. Ultrastructure of Hendra virus and Nipah virus within cultured cells and host animals. Microbes Infect. 2001;3:297-306 pubmed
- Drosten C, Gunther S, Preiser W, van der Werf S, Brodt H, Becker S, et al. Identification of a novel coronavirus in patients with severe acute respiratory syndrome. N Engl J Med. 2003;348:1967-76 pubmed
- Reed K, Melski J, Graham M, Regnery R, Sotir M, Wegner M, et al. The detection of monkeypox in humans in the Western Hemisphere. N Engl J Med. 2004;350:342-50 pubmed
- Fauquet, C. M., M. A. Mayo, J. Maniloff, U. Desselberger, and L. A. Ball (ed.). 2005. Virus taxonomy. Classification and nomenclature of viruses. Eighth report of the International Committee on the Taxonomy of Viruses. Elsevier Academic Press, New York, NY.
- unpublished data.
- 来邦网
- 英文来邦